化学構造式は平面(2D)で描かれますが、実際の分子の形は立体的(3D)です。 同じ平面構造でも3Dの形が異なる場合があり、これを「異性体」と呼びます。 異性体は形が違うと性質も異なるため、分子を3Dでとらえることは重要です。
今回は、Pythonで、目には見えない分子の3D構造をパソコン上で動かしながら観察します。さらに分子の異性体についても見ていきます!
1. なぜ分子を立体でとらえることが大切なのか?
分子式は同じなのに構造が違うものを異性体と呼びます。
今回は、側鎖は同じで、構造が異なる立体異性体の中で、鏡像異性体について触れてみます。
鏡像異性体とは、鏡に映した左手と右手のような関係の構造のこと言います。
例えば、乳酸では、構造を平面構造で書く場合と立体構造で書く場合を示します。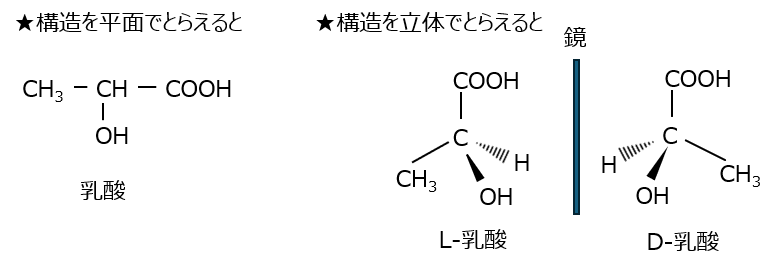
※L体とD体の決定は、フィッシャー投影式の情報を参考にされてください。
このように、平面でとらえると同じでも、立体的に表示すると重ならない、異なる構造も存在することが分かります。
📢分子式は同じなのに立体構造が違うだけで、性質が違う例
・薬の例(サリドマイド)
サリドマイド(C13H10N2O4)は鎮静剤や、妊婦のつわりを軽減する薬として広く使用されていました。しかし、この薬を妊娠初期に服用した母親から生まれた多くの胎児に、手足が短くなるなどの深刻な奇形が生じる被害がありました。薬として販売されていたサリドマイドには、R体とS体という2種類の鏡像異性体が混ざっていました。後の研究で、望ましい鎮静作用を持つのはR体であり、一方で胎児に深刻な奇形を引き起こすのはS体であることが明らかになり、薬は販売中止となりました。このR体とS体は、体の中に入ると、お互いの形に簡単に変化し合う性質(ラセミ化)があることが分かってきてます。人間にとって安全かつ効果のあるR体だけを使用するのは難しい、というのが現状のようです。
※R体とS体の決定は、RS表記法の情報を参考にされてください。
・香りの例(リモネン)
柑橘系の香りの成分リモネン(C10H16)。これも鏡像異性体があり、一方(D体)はオレンジ 🍊 の香り、もう一方(L体)はレモン 🍋 の香りとして、私たちの鼻は認識し分けているのです!
立体構造をとらえて初めて分かる事実です。
2. Pythonで分子の3Dモデルを作ってみよう!
Pythonを使って3Dモデルを作ってみます!今回も、Google Colaboratoryを使って進めていきましょう。
Step 1:ライブラリの準備
化学情報を扱うためのRDKitと、3D表示を行うためのpy3Dmol、化学物質名を検索するためのPubChemPyの3つのライブラリをインストールします。Colabの新しいセルに、以下のコードを貼り付けて実行してください。
# ライブラリのインストール !pip install rdkit -q !pip install py3Dmol -q !pip install pubchempy -q
Step 2:SMILESから3D構造を生成する(スティックモデル)
次に、私たちの作業を簡単にする2つの関数をPythonに覚えさせます。
・get_smiles:物質名(例: ‘methane’)をPubChemで検索し、SMILESコードを取得する関数。
・show_3d:SMILESコードから3D構造を計算し、py3Dmolで表示する関数。
以下のセルを実行すると、コンピュータがこれら2つの命令を覚えた状態になります。(このセル自体は何も表示しません)
# ===== 必要なライブラリのインポート =====
from rdkit import Chem
from rdkit.Chem import AllChem
import py3Dmol
import pubchempy as pcp
from IPython.display import display, HTML
# ===== 関数1:PubChemからSMILESを取得する関数 =====
def get_smiles(chemical_name):
print(f"PubChemで「{chemical_name}」を検索中...")
try:
# PubChemで名前を検索
compounds = pcp.get_compounds(chemical_name, 'name')
# ▼ チェック1:化合物が見つからなかったら、ここで終了
if not compounds:
print("エラー: 化合物が見つかりませんでした。")
return None # Noneを返して関数を抜ける
# ▼ チェック2:SMILESコードが空だったら、ここで終了
smiles = compounds[0].smiles
if not smiles:
print("エラー: SMILESコードが見つかりませんでした。")
return None # Noneを返して関数を抜ける
# ▼ すべてのチェックを通過したら、成功
print(f"SMILESコードを取得しました: {smiles}")
return smiles
except Exception as e:
print(f"検索中にエラーが発生しました: {e}")
return None
# ===== 関数2:SMILESから3Dモデルを表示する関数 =====
def show_3d(smiles, style='stick'):
if not smiles:
print("SMILESコードが有効でないため、表示できません。")
return
try:
# ===== SMILESから3D構造を生成 =====
mol = Chem.MolFromSmiles(smiles)
if mol is None:
print(f"エラー: RDKitがSMILES「{smiles}」を認識できませんでした。")
return
mol = Chem.AddHs(mol) # 水素原子を付加
AllChem.EmbedMolecule(mol) # 3D構造を生成
AllChem.MMFFOptimizeMolecule(mol) # 構造を最適化
mol_block = Chem.MolToMolBlock(mol)
# ===== py3Dmolで3D表示 =====
view = py3Dmol.view(width=500, height=400)
view.addModel(mol_block, 'mol')
# 表示スタイルを設定
if style == 'stick':
view.setStyle({'stick':{}})
elif style == 'sphere':
# 原子を球(sphere)で、結合を棒(stick)で表示
view.setStyle({'stick':{'radius': 0.1}, 'sphere':{'scale': 0.3}})
view.zoomTo()
view.show() # Colab上に表示
except Exception as e:
print(f"3Dモデルの生成中にエラーが発生しました: {e}")
print("関数の定義が完了しました。次のセルから3Dモデルを作成できます。")
Step 3:最初の3Dモデル!「メタン」を名前で表示しよう
早速、一番シンプルなメタン(CH₄)の形を見てみましょう。
さきほど準備したget_smiles関数とshow_3d関数を組み合わせて使います。
# 1. 'methane' という名前でSMILESコードを取得
smiles_methane = get_smiles('methane')
# 2. 取得したSMILESを使って3D表示
if smiles_methane:
show_3d(smiles_methane)
## 出力結果 PubChemで「methane」を検索中... SMILESコードを取得しました: C
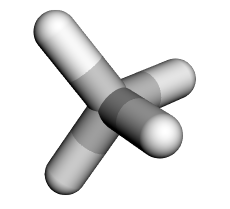
セルの下に3Dモデルが表示され、マウスでぐりぐり動かせるはずです! 炭素原子を中心に、4つの水素原子が正四面体の頂点方向に伸びています。
Step 4:表示スタイルを変えてみよう(球棒モデル)
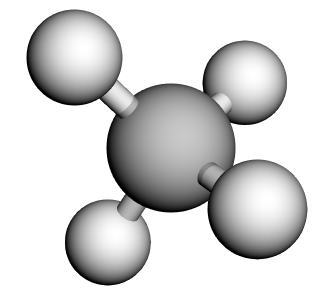
show_3d関数は、表示スタイルを変えられます。原子を球で表す球棒(きゅうぼう)モデルで表示してみます。
## 2番目の引数でスタイルを 'sphere' に指定
if smiles_methane:
show_3d(smiles_methane, style='sphere')
3.【探究】いろいろな異性体を立体的に見てみよう!
異性体の「形」の違いを、自分の手で確かめていきましょう。
💡探究1:構造異性体(ブタン vs イソブタン)
分子式はどちらもC4H10ですが、炭素骨格が違います。
# ブタン(直鎖状)
smiles_butane = get_smiles('butane')
if smiles_butane:
print("--- ブタン (n-Butane) ---")
show_3d(smiles_butane, style='sphere')
# イソブタン(分岐状)
smiles_isobutane = get_smiles('isobutane')
if smiles_isobutane:
print("--- イソブタン (Isobutane) ---")
show_3d(smiles_isobutane, style='sphere')
PubChemで「butane」を検索中... SMILESコードを取得しました: CCCC --- ブタン (n-Butane) --- PubChemで「isobutane」を検索中... SMILESコードを取得しました: CC(C)C --- イソブタン (Isobutane) ---
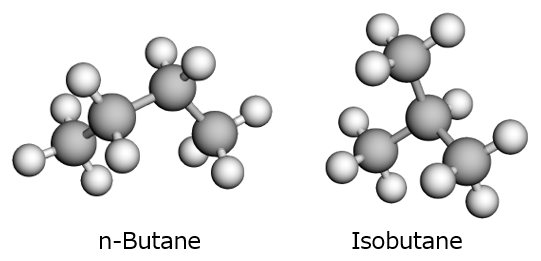
それぞれのモデルを動かして、形の違いを観察してみてください。ブタンは細長く、イソブタンは球に近い形をしています。この形の違いが、分子同士のくっつきやすさに関係し、沸点の違い(ブタン: -0.5℃, イソブタン: -11.7℃)を生み出しています。
💡探究2:鏡像異性体(リモネン)
先に紹介した、オレンジ 🍊 とレモン 🍋 の香りの違いを生むペアです。 PubChemで「D-limonene」「L-limonene」と名前を指定して検索してみましょう。
# D-リモネン (オレンジの香り)
smiles_d_limonene = get_smiles('D-limonene')
if smiles_d_limonene:
print("--- D-リモネン (オレンジの香り) ---")
show_3d(smiles_d_limonene)
# L-リモネン (レモンの香り)
smiles_l_limonene = get_smiles('L-limonene')
if smiles_l_limonene:
print("--- L-リモネン (レモンの香り) ---")
show_3d(smiles_l_limonene)
# 出力 PubChemで「D-limonene」を検索中... SMILESコードを取得しました: CC1=CC[C@@H](CC1)C(=C)C --- D-リモネン (オレンジの香り) --- PubChemで「L-limonene」を検索中... SMILESコードを取得しました: CC1=CC[C@H](CC1)C(=C)C --- L-リモネン (レモンの香り) ---
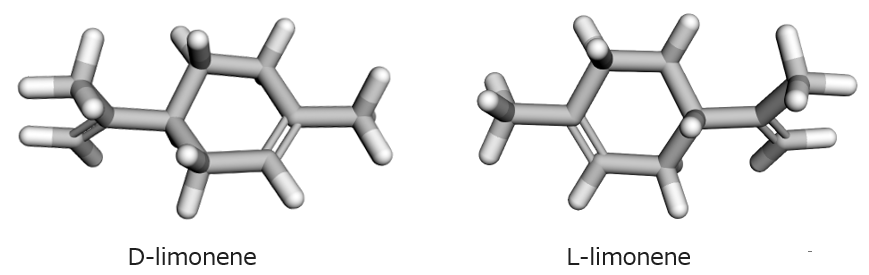
一見、同じように見えますが、マウスで回転させて、絶対に重ね合わせることができないことを確かめてみてください。これが鏡像異性体です。私たちの鼻は、この微妙な「形」の違いをちゃんとかぎ分けています。
4. まとめ
今回は、Pythonを使って分子の立体的な「形」を可視化する方法を学びました。
・PubChemPy は、物質名からSMILESコードを検索できる。
・RDKit は、SMILESから3D構造を計算できる。
・py3Dmol は、その3Dモデルをインタラクティブに表示してくれる。
・異性体の立体的な形の違いを確認した。
分子の形が、薬の効果や香りの違いに繋がります。
他にも、下記のような取り組みなどを通じて、化学の”探究”をしてみてください!
・身近な薬(例: ‘Aspirin’, ‘Ibuprofen’, ‘Paracetamol’)の3D構造を調べてみよう。
・糖類(例: ‘Glucose’, ‘Fructose’)の形を比べてみよう。
・自分の好きな香り(例: ‘Vanillin’ (バニラ), ‘Menthol’ (ミント))の形を見てみよう。


コメント