このカテゴリーの記事では、「Pythonを使った機械学習やケモ・バイオインフォマティクスの実装や論文」を紹介していきたいと考えています。Python は3系(3.7)、anacondaを中心にして環境構築していきます。以下のようなハード・ソフト環境(CentOS Linux)を用いますが、Python(anaconda)が利用できれば、異なる環境下でも類似の実装が可能かと思います。
- CPU Intel® Core™ i7-6700K CPU @ 4.00GHz × 8
- メモリ 64GB
- GPU GeForce GTX 1080/PCIe/SSE2
- OS CentOS Linux 8
- Python 3.7
はじめに
前回の記事で、conda を利用して、BLASTによる相同性検索を実行できる Local な仮想環境を構築しました。また、データベースとして、ヒト, Homo sapiensの全タンパク質(推定の物も含む)の一次構造をダウンロードし、検索可能な状態にしました。

今回の記事では、この環境を使って、多種類のタンパク質を問合せ配列(Query(クエリ))としたBLAST検索を行い、結果を分析してみたいと思います。
クエリとしてのタンパク質一次構造のダウンロード
(1)出芽酵母Saccharomyces cerevisiaeの全タンパク質
まず、例として、酵母(イースト)として食品や酒類の発酵にも用いられる、出芽酵母 Saccharomyces cerevisiae の全てのタンパク質(推定の物も含む)を問合せ配列(クエリ)としたBLAST検索を行ってみます。そのために、Saccharomyces cerevisiae S288C 株の全タンパク質の一次構造をダウンロードします。前回も利用した NCBI のサイトからダウンロードすることができます。https://www.ncbi.nlm.nih.gov/genome/15 の protein と書かれたリンクをクリックするか、下記のコマンドを実行してください。前回用意した仮想環境 blast をアクティブにし、human_protein ディレクトリにダウンロードします。
$ conda activate blast
$ cd /home/xxxx/human_protein #xxxxの箇所は適宜修正してください。
$ wget https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/146/045/GCF_000146045.2_R64/GCF_000146045.2_R64_protein.faa.gz
これで圧縮形式としてダウンロードされますので、さらに以下のように解凍してください。
$ gzip -d GCF_000146045.2_R64_protein.faa.gz
GCF_000146045.2_R64_protein.faaというファイルが得られるはずです。このテキストファイルに fasta 形式で、全てのタンパク質の一次構造が羅列されています。このファイルを指定する形で、次のコマンドを実行してみて下さい。
$ grep -c '>' GCF_000146045.2_R64_protein.faa
” 6002 ” という出力が得られたと思います(2021年3月現在)。grep は 特定の文字列を含むファイルやその行を表示させるコマンドで、通常のLinuxであれば使用可能です。Windows 等をお使いの場合には、テキストを表示させ、” > ” という文字を検索してください。6002個あると思います。fasta 形式では、> の後にそれぞれのタンパク質(遺伝子)の番号や名前等をつけ、改行後、アミノ酸(塩基)配列を記すことになっています。この ” > ” を数え上げることで、Saccharomyces cerevisiae S288C株の全タンパク質(推定を含む)の数が6002個であることがわかりました。
(2)チンパンジーの1番染色体タンパク質
2つ目の例として、チンパンジー(黒猩猩、Pan troglodytes) のタンパク質(推定の物も含む)を問合せ配列(クエリ)としたBLAST検索も行ってみたいと思います。全てのタンパク質で検索を行うと、少し時間がかかりますので、遺伝子が1番染色体に存在するタンパク質についてのみダウンロードし、使用したいと思います。UniProt のこちらhttps://www.uniprot.org/proteomes/UP000002277のページからダウンロードできます。Chromosome 1 の左横の□にチェックを入れて、すぐ上のDownloadボタンをクリックし、Goをクリックすることでダウンロード可能です。uniprot-proteome_UP000002277+AND+(proteomecomponent__Chromosome+1_).fasta.gz という圧縮ファイルが得られますので、下記コマンドで解凍してください。
$ gzip -d 'uniprot-proteome_UP000002277+AND+(proteomecomponent__Chromosome+1_).fasta.gz'
UniProt のページにも表示されていますが、解凍で得られたファイルに対して、先程と同様のgrep コマンドを実行することで、チンパンジーの1番染色体には4550個のタンパク質がコードされていることがわかります。
BLAST検索の実行
(1)出芽酵母Saccharomyces cerevisiaeの全タンパク質をクエリとした検索
まずは、出芽酵母のタンパク質6002個をクエリとしてBLAST検索を行ってみたいと思います。前回の記事では、マウスのミオグロビンという1つのタンパク質で検索を行いましたが、同様のコマンドで、一度に検索を実行することが可能です。仮想環境 blast をアクティブにした状態で、以下を実行してください。
$ blastp -query GCF_000146045.2_R64_protein.faa -db GCF_000001405.39_GRCh38.p13_protein.faa -out yeast.out -num_threads 8 -evalue 0.001 -num_alignments 1
この例では、出芽酵母のタンパク質6002個(GCF_000146045.2_R64_protein.faa)がクエリで、-query の後ろにファイル名を指定しています。-db GCF_000001405.39_GRCh38.p13_protein.faaは前回用意したヒトの全タンパク質の配列データベースを指定しています、また、-out の後ろに、検索結果の保存ファイル名をyeast.out と指定しています。-num_threads 8 はCPUコア数を指定していますので、コンピュータに応じて変更して下さい。-evalue 0.001 の部分では、Expect (E) value の閾値を 0.001に指定しています。この設定で、E value が0.001以下の検索結果のみを得ることができ、これ以上の値を示す検索結果は、ヒット無しとして扱うことができます。E value は偶然に同程度の類似性スコアを示す配列が得られる期待値(本数)を表しており、この値が小さい程、その検索結果は偶然の一致ではないことを示しています。詳細はこちらやこちら等を確認してください。
6002個のBLAST検索が数十分で完了します。得られた yeast.out ファイルはテキストファイルで、以下のような結果が書き込まれていると思います。
・
・
・
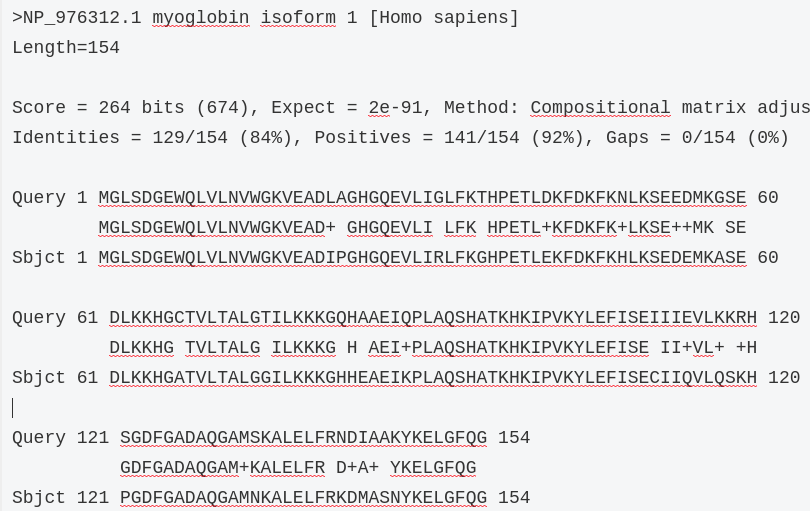
Query= NP_001018029.1 Coq21p [Saccharomyces cerevisiae S288C]
Length=66
***** No hits found *****
Lambda K H a alpha
0.319 0.121 0.371 0.792 4.96
Gapped
Lambda K H a alpha sigma
0.267 0.0410 0.140 1.90 42.6 43.6
Effective search space used: 1885343980
Query= NP_001018030.1 L-serine/L-threonine ammonia-lyase CHA1
[Saccharomyces cerevisiae S288C]
Length=360
Score E
Sequences producing significant alignments: (Bits) Value
NP_006834.2 L-serine dehydratase/L-threonine deaminase [Homo sapi... 145 6e-40
XP_011536148.1 serine dehydratase-like isoform X2 [Homo sapiens] 138 3e-37
XP_005253888.1 serine dehydratase-like isoform X2 [Homo sapiens] 138 3e-37
NP_612441.1 serine dehydratase-like [Homo sapiens] 138 3e-37
NP_001291922.1 serine dehydratase-like [Homo sapiens] 138 3e-37
XP_016874252.1 serine dehydratase-like isoform X1 [Homo sapiens] 139 4e-37
XP_011522276.1 serine racemase isoform X1 [Homo sapiens] 56.6 4e-08
XP_006721629.1 serine racemase isoform X1 [Homo sapiens] 56.6 4e-08
XP_006721628.1 serine racemase isoform X1 [Homo sapiens] 56.6 4e-08
NP_068766.1 serine racemase isoform a [Homo sapiens] 56.6 4e-08
・
・
・
6002個のうち、1本目の Query= NP_001018029.1 Coq21p は、ヒトの全タンパク質に対するBLAST検索でヒット無し(No hits found)です。即ち、出芽酵母が有するこのタンパク質を、ヒトは持っていない可能性があります(*ここで E value の値が問題になります。即ち、どれ程の類似性があるときに、ヒットとするかの問題があります。今回の結果だけを鵜呑みにすることはできませんが、類似性の高いタンパク質がヒトには存在しないのは間違いないはずです。)。また、2本目の Query= NP_001018030.1 L-serine/L-threonine ammonia-lyase CHA1 については、非常に小さい E value の値を示す検索結果が得られており、一定程度の類似性を示すタンパク質(ヒトの場合、L-serine dehydratase/L-threonine deaminase と名付けれられている)がヒトにも存在すると言ってよさそうです。
次に、検索結果 yeast.out に対して、以下のコマンドを実行してみてください。
$ grep -c 'No hits found' yeast.out
今回のBLAST検索設定において、ヒット無し(No hits found)となったタンパク質の数を数えています。手元の結果では、” 2552 ” という結果が得られました。このことから、2552 / 6002 * 100 % ≒ 約 43% のタンパク質がヒトには存在しない(高い類似性を示さない)ということが推定できました。逆に考えれば、出芽酵母のタンパク質のうち、57%程度は、ヒトにも類似した(相同である可能性のある)タンパク質が存在することになります。
(2)チンパンジーの1番染色体タンパク質をクエリとした検索
チンパンジーのタンパク質の一部(1番染色体にコードされている4550個)についても、同様の検索をやってみましょう。以下を実行してください。
$ blastp -query 'uniprot-proteome_UP000002277+AND+(proteomecomponent__Chromosome+1_).fasta' -db GCF_000001405.39_GRCh38.p13_protein.faa -out Ch1.out -num_threads 8 -evalue 0.001 -num_alignments 1
検索結果を Ch1.out ファイルで保存するようにし、出芽酵母の場合と同様の設定でBLAST検索しました。こちらの場合も数十分あれば、完了します。先程と同様にヒット無し(No hits found)を数えてみましょう。
$ grep -c 'No hits found' Ch1.out
チンパンジーの場合には、” 96 ” という結果が得られます。即ち、96 / 4550 * 100 % ≒ 約 2.1% のタンパク質がヒトには存在しない(高い類似性を示さない)ということが推定できました。

逆に考えれば、チンパンジーでは、1番染色体にコードされているタンパク質の98%程度は、ヒトにも類似の(相同な)タンパク質が存在することになりますね。
まとめ
今回、酵母とチンパンジーの合計で1万種類程度のタンパク質をクエリとして、ヒトのタンパク質データベースに対してBLAST検索を実施してみました。酵母とヒト、チンパンジーとヒト、それぞれの遺伝子・タンパク質の類似性について、BLAST検索結果がヒットするかどうかで推定してみました。以下の記事では、今回得られた検索結果について、どの程度の類似性を示すタンパク質がどのくらい存在するのか、それらはどのようなタンパク質なのか等、Jupyter notebookを利用して、もう少し解析しています。


コメント