このカテゴリーの記事では、「Pythonを使った機械学習やケモ・バイオインフォマティクスの実装や論文」を紹介していきたいと考えています。Python は3系(3.7)、anacondaを中心にして環境構築していきます。以下のようなハード・ソフト環境(CentOS Linux)を用いますが、Python(anaconda)が利用できれば、異なる環境下でも類似の実装が可能かと思います。
● CPU Intel® Core™ i7-6700K CPU @ 4.00GHz × 8
● メモリ 64GB
● GPU GeForce GTX 1080/PCIe/SSE2
● OS CentOS Linux 8
● Python 3.7
はじめに
前回の記事に引き続き、BLAST検索結果の分析を進めていきます。データ解析用ライブラリ Pandas や Jupyter notebook の詳細については、参考になるwebサイトが多数ありますので、それらも参考にしてください。
例えば、
● Pandas について note.nkmk.me さま https://note.nkmk.me/pandas/
● Jupyter notebook について Udemy さま https://udemy.benesse.co.jp/development/python-work/jupyter-notebook.html
前々回の記事で、出芽酵母の全タンパク質 6002個をクエリとしたBLAST検索を行い、3450個のタンパク質は、ヒトの何らかのタンパク質にヒットし、E value 0.001以下を示す類似タンパク質がヒトに存在することがわりました。前回の記事では、検索結果を保存したyeast.out ファイルから、タンパク質一次構造同士の一致割合を示す、Identities(%)の情報を抽出し、Identities.out として保存しました。この情報は、1列、3450行のデータとみなすことができます。前回インストールした、Jupyter notebookを使って、Pandas や Plotly 等で分析してみます。

前回、前々回の記事は下記を参照してください。
Jupyter notebook の新規作成
まず、仮想環境 blastをアクティブにし、Jupyter notebook を立ち上げましょう。
$ cd /home/xxxx/human_protein $ conda activate blast $ jupyter notebook
上記ブラウザが起動します。
新規をクリックし、Python 3 を選択します。
以下のように、新しい Untitled のノートブックが作成できました。
このノートブックに(1)セル毎にPython のコマンドを記述し、(2)実行することができ、その出力を確認しながら、分析を進めていくことができます。では実際に、Identities.out を読込み、分析していきましょう。
Pandas, Plotlyによる分析
まずは、以下を記述して、Run します。
import pandas as pd
import plotly.express as px
これは、前回導入した、PandasとPlotlyというライブラリを使うために必要な、おまじないと考えて下さい。次に以下をRunします。
df = pd.read_table('./Identities.out', names=('Identity',))
この指令で、Pandasライブラリの read_table というメソッドを使って、Identities.out に抜き出した一致割合(%)の値(3450個分)を pandas.DataFrame という形式で読み込みました。その際に、names = (‘Identity’,) と指定することで、列にIdentity という名前を付けました。さらに df = ・・・ と記述することで、それを変数 df としています。
次のように、単に df を実行すると、そのDataFrame を表示することができます。
df
0行目から3449行目まで、一致割合(%)が読み込まれていることがわかります。
ここで、( XX% )と表示されているように、今回読み込んだ値は、それぞれが object 型 で読み込まれています。このままでは、これらの値に四則演算を適用することができません。そのため、以下のセルを実行し、()と%を削除した上で、object 型を数値型へと変更します。
df['Identity'] = df['Identity'].str.strip('(, %, )')
df = df.apply(pd.to_numeric)
df
このようになりました。
これで、様々な数値計算が可能です。Pandasライブラリには、色々なメソッド・関数が用意されており、下記のように変数 df に対して .describe() を実行するだけで、平均値、標準偏差、最小・最大値、中央値、四分位数などの要約統計量を計算できます。
print(df.describe())
一致割合の平均値(mean)は 38(%) と計算されました。また、最大値(max)が 96(%) と表示されており、非常に一致割合(%)が高いタンパク質も存在することがわかります。
つぎに、以下のメソッドを実行してみましょう。
print(df['Identity'].value_counts())
このように、3450個の検索結果について、一致割合(%)毎にタンパク質の数を数え上げることができました。30%(158個が該当)、28%(156個が該当)、29%(150個が該当)・・・の順番に多いことがわかります。平均値は38%であり、分布に偏りがあるようです。
そこで、この結果を Plotly を用いてヒストグラムにしてみましょう。下記のセルを実行してください。
fig = px.histogram(data_frame = df, x = 'Identity')
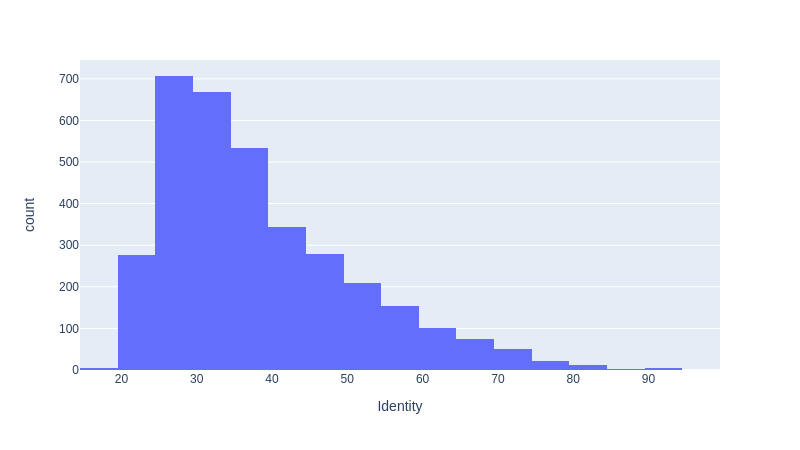
fig
ヒットしたタンパク質3450個について、一致割合(%)ごとにカウント・プロットしたヒストグラムが得られました。Plotlyで書いたグラフでは、マウスポインタを合わせることで、プロット点に関する情報が表示されます。また、図の右上にあるアイコンを使って、拡大・縮小や軸範囲の変更等自由自在に行うことができるなど、高機能なグラフを描くことができます。
ヒストグラムは、横軸の右側(高割合側)に裾が広いプロットとなっており、酵母のタンパク質には、ヒトのタンパク質と非常に高い類似性を有するものが少数存在することがわかります。例えば、グラフ右端にマウスを合わせれば、96% と非常に高い一致割合を示すタンパク質が1個だけ存在することがわかります。これはユビキチン というタンパク質で、生物種間で一次構造および立体構造の保存性が非常に高いことが知られています。
このヒストグラムは、下記のように設定を変更することで、5%毎にカウント数をまとめることができます。
fig2 = px.histogram(data_frame=df, x = 'Identity', nbins=20)
fig2
カウント数を5%刻みでまとめることで、右側に広がった分布の傾向がより明確になったかと思います。数行のコードを実行するだけで、BLAST検索結果の統計的な分析とグラフ化が出来ました。

このJupyter notebookを保存しておけば、データが変わってもグラフ化まで、数十秒で完了することができます。勿論、BLAST検索以外の様々な解析結果について、このような分析が可能ですので、今後も少しずつ紹介したいと思います。



コメント