このカテゴリーの記事では、「Pythonを使った機械学習やケモ・バイオインフォマティクスの実装や論文」を紹介していきたいと考えています。Python は3系(3.7)、anacondaを中心にして環境構築していきます。以下のようなハード・ソフト環境(CentOS Linux)を用いますが、Python(anaconda)が利用できれば、異なる環境下でも類似の実装が可能かと思います。
● CPU Intel® Core™ i7-6700K CPU @ 4.00GHz × 8
● メモリ 64GB
● GPU GeForce GTX 1080/PCIe/SSE2
● OS CentOS Linux 8
● Python 3.7
はじめに
以下の記事で、分子動力学法(Molecular dynamics)の実装として、OpenMM のインストールとテストを実行しました。
分子動力学計算によって、通常の実験では検証が困難な、分子内・間の相互作用や運動が関係する様々な過程をシミュレーションすることができます。今後、公式のチュートリアルや例題を紹介していきますが、まず一般的な例題として、Trp-cageを例に、タンパク質の折りたたみ(Protein Folding)シミュレーションをOpenMM で行ってみたいと思います。
Trip-cage はエクセンディン 4 というタンパク質の C 末端側半分をもとに設計された、アミノ酸20残基からなる小さいタンパク質であり、その立体構造も決定されています(PDB ID:1L2Y)。このタンパク質は6番目の残基がトリプトファン(Trp)であり、その大きな疎水性側鎖が、分子の中央にすっぽりと収納されたような構造を形成するため、Trp-Cageとよばれています。また、20残基と小さいこともり、分子動力学シミュレーションによる研究対象としても有名です。分子動力学シミュレーションの文献としては、下記などを参考にして下さい。
● J Am Chem Soc 2002 Sep 25;124(38):11258-9. doi: 10.1021/ja0273851. All-atom structure prediction and folding simulations of a stable protein Carlos Simmerling 1, Bentley Strockbine, Adrian E Roitberg
● PNAS November 11, 2003 100 (23) 13280-13285; https://doi.org/10.1073/pnas.2233312100 Trp-cage: Folding free energy landscape in explicit water Ruhong Zhou
● Chemistry Letters 2021, Vol.50, No.1 162-165 The Folding of Trp-cage is Regulated by Stochastic Flip of the Side Chain of Tryptophan Takunori Yasuda, Yasuteru Shigeta, and Ryuhei Harada https://doi.org/10.1246/cl.200699
まず本記事では、分子グラフィックスツール PyMOLを用いて、折りたたむ前の(アンフォールド状態の)Trp-cageの構造を構築したいと思います。本記事の方法を使えば、他のペプチドやタンパク質についても同様な構造モデリングが可能になります。PyMOLのインストールについては、以下の記事も参考にして下さい。
PyMOLによる分子構造の構築
分子構造の構築を順番にみていきますが、以下の動画に要点をまとめていますので参考にしてください。
前回、OpenMMのインストールを行った際、同じ仮想環境 openmm にPyMOLもインストールしました。ですので、以下のようにopenmmをアクティブにした後、pymol コマンドで起動することができます。
$ cd OpenMM #OpenMMディレクトリに移動(別のディレクトリでもよい。) $ conda activate openmm #仮想環境openmm をアクティブに $ pymol #PyMOLを起動
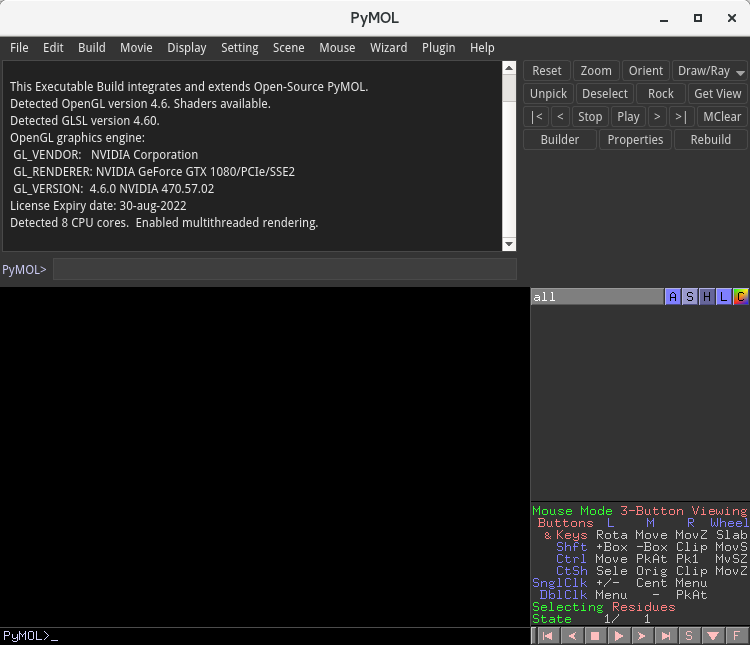
上のように PyMOL ウインドウが起動するはずです。PyMOLは、pdbファイルや電子密度ファイルを読み込み、構造の観察・分析や、図・動画を書き出したりといった用途で使用されることが多いです。一方、Build 機能を使うことで、分子構造を一から構築することも可能です。以下で、Trp-cage の構造(フォールディング前の伸びた構造)を構築してみましょう。
ここで、Trp-cage の一次構造、即ちアミノ酸配列はわかっている必要があります。こちらのページ等に記載されているように、「NLYIQWLKDGGPSSGRPPPS」です。三文字表記では、Asn-Leu-Tyr-Ile-Gln-Trp-Leu-Lys-Asp-Gly-・・・です。6番目のアミノ酸がトリプトファン(W, Trp)になっていますね。このアミノ酸配列に従うように、N末端からアミノ酸をつなげる必要があります。
まず、PyMOLウインドウの上部のメニュー一覧の「Build」をクリックし、Buildメニューを表示してください。さらに、そのメニューのなかから、「Residue」にマウスカーソルを合わせてください。
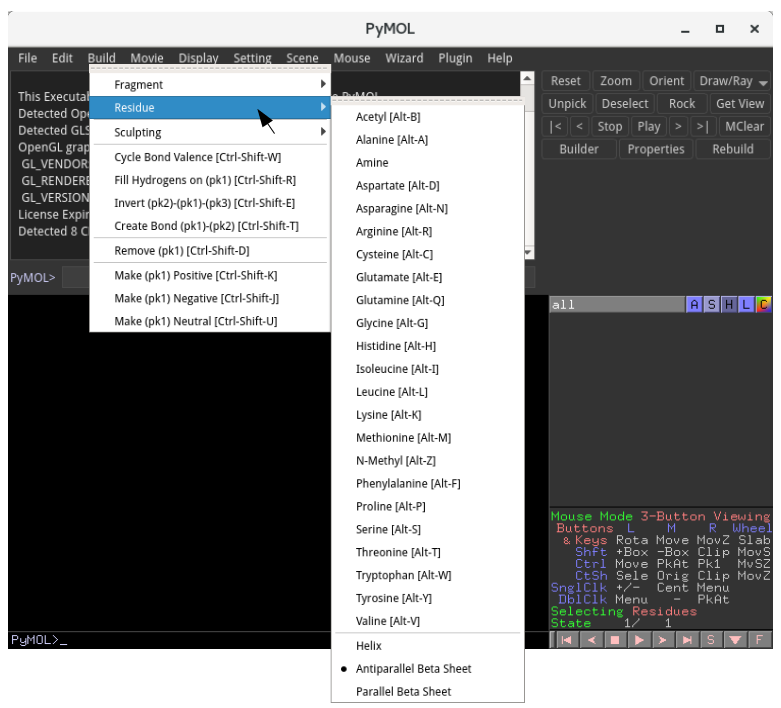
上記のようなウインドウが表示され、アミノ酸残基等のリストを選択可能になります。1番目のアミノ酸はアスパラギン(ASN)ですので、Asparagine [Alt-N] を選択してください。
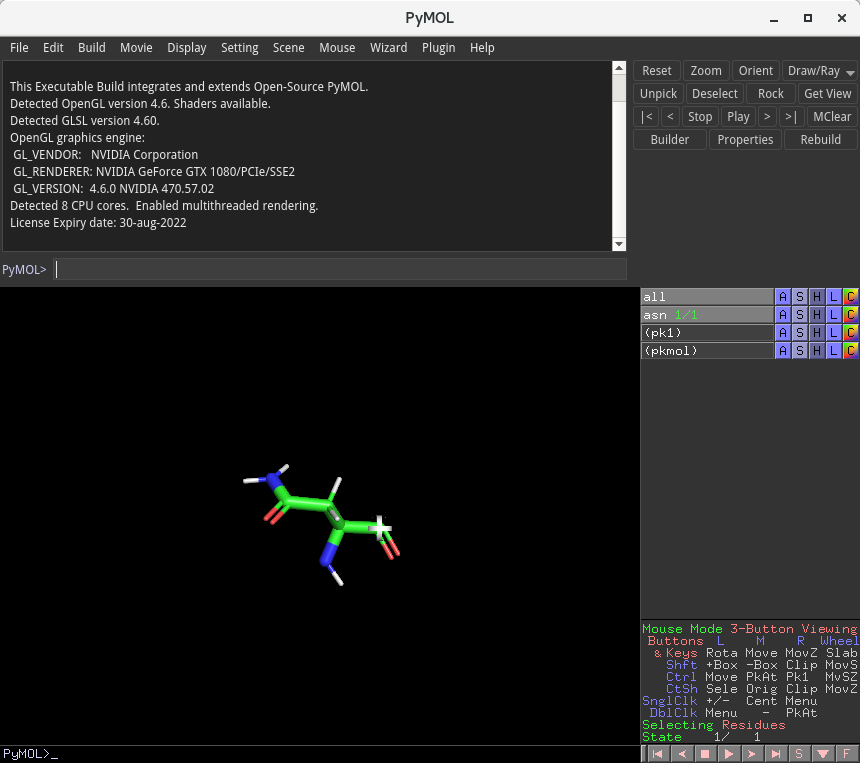
上のように、アスパラギンのモデルが表示されると思います。以降は、この操作(Build → Residue → アミノ酸を選択)を繰り返します。即ち、2番目のアミノ酸はロイシン(L, Leu)ですので、以下のように、Build → Residue → Leucine と選択します。
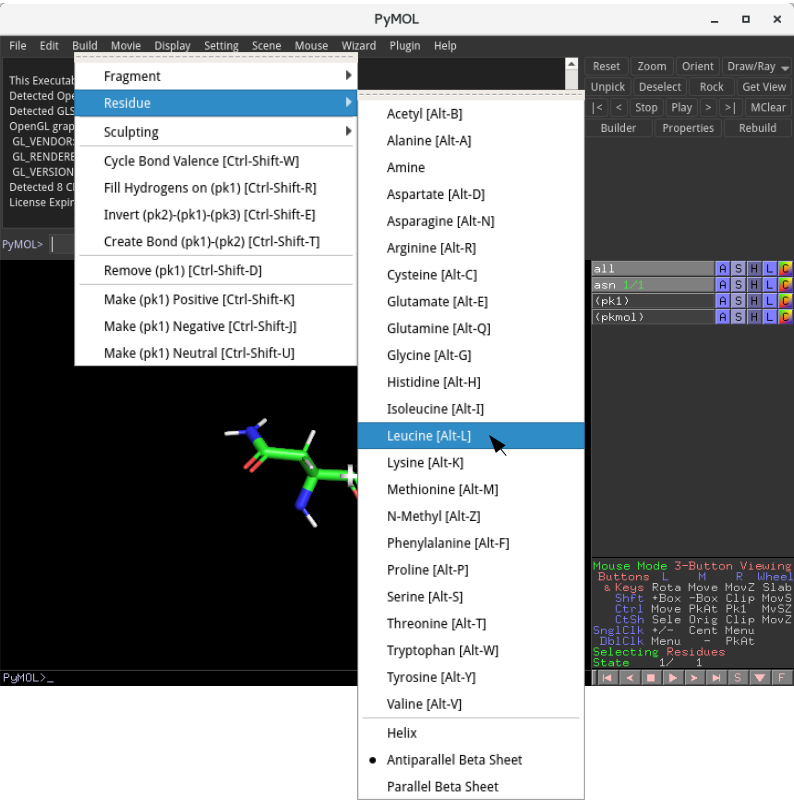
これで以下のようになるかと思います。
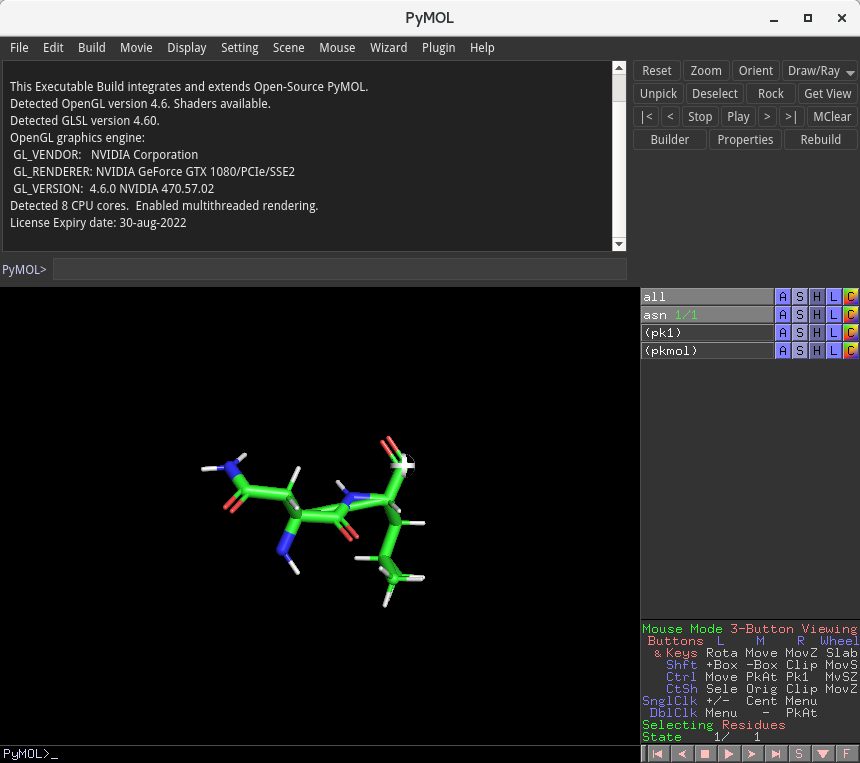
Asn-Leu という2個のアミノ酸からなるべプチドの分子構造が構築されました。これを残りの18個のアミノ酸について繰り返すことで、各アミノ酸をペプチド結合で連結していくことが可能です。
また、アミノ酸選択ウインドウにおいて、Antiparallel Beta Sheet が●で選択されていることが確認できると思います。このままの設定で20残基を結合させると、主鎖構造が真っ直ぐ同じ向きに伸びきった状態となり、以下のようにある方向にだけ長い構造となってしまいます。
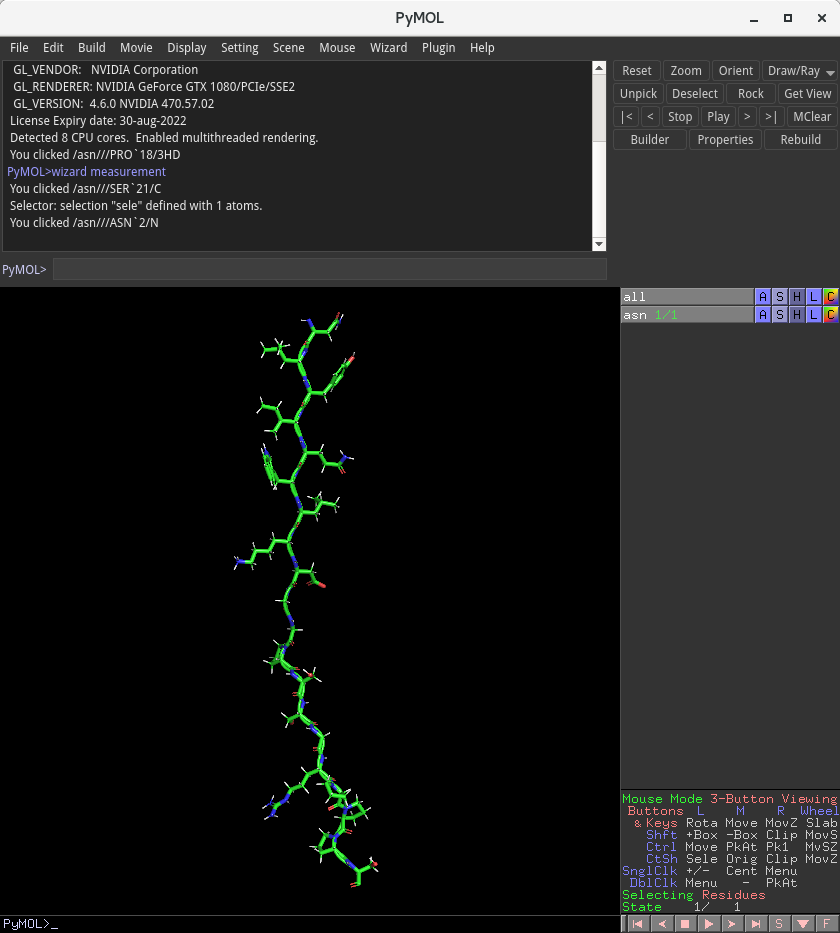
これはこれで、アンフォールドした状態の構造として誤りというわけではありませんが、どこまでもまっすぐに伸びきっているのは不自然な感じがします。また、1軸方向にのみ長い構造は、分子動力学シミュレーションの設定上も適していません。
そこで、5、6残基に1回程度、Antiparallel Beta Sheetのひとつ上にある、Helixを選択し、その後でアミノ酸を指定してみます。この際、次の残基を選択する際には、まず設定をAntiparallel Beta Sheetへと戻すようにしてください。
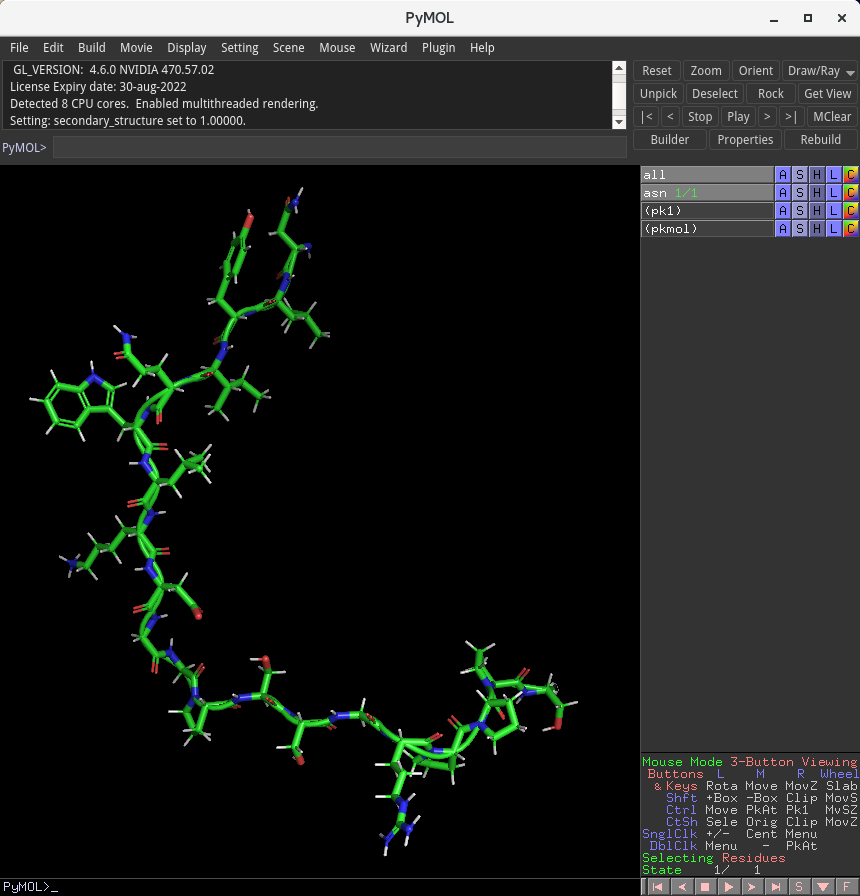
6番目のW、11番目のG、17番目のPをつなぐ際にHelixを選択することで、上のように少し曲った構造を構築することができました。これを分子動力学シミュレーションの初期構造としたいと思います。
また最後に、C末端のカルボキシル基酸素が不足していますので、それを追加したいと思います。下記のようにBuilderメニューをクリック、起動してください。
Builderウインドウが出現しました。C末端の20番目セリンを画面中央にすると、カルボニル炭素に水酸基が不足していることがわかると思います。
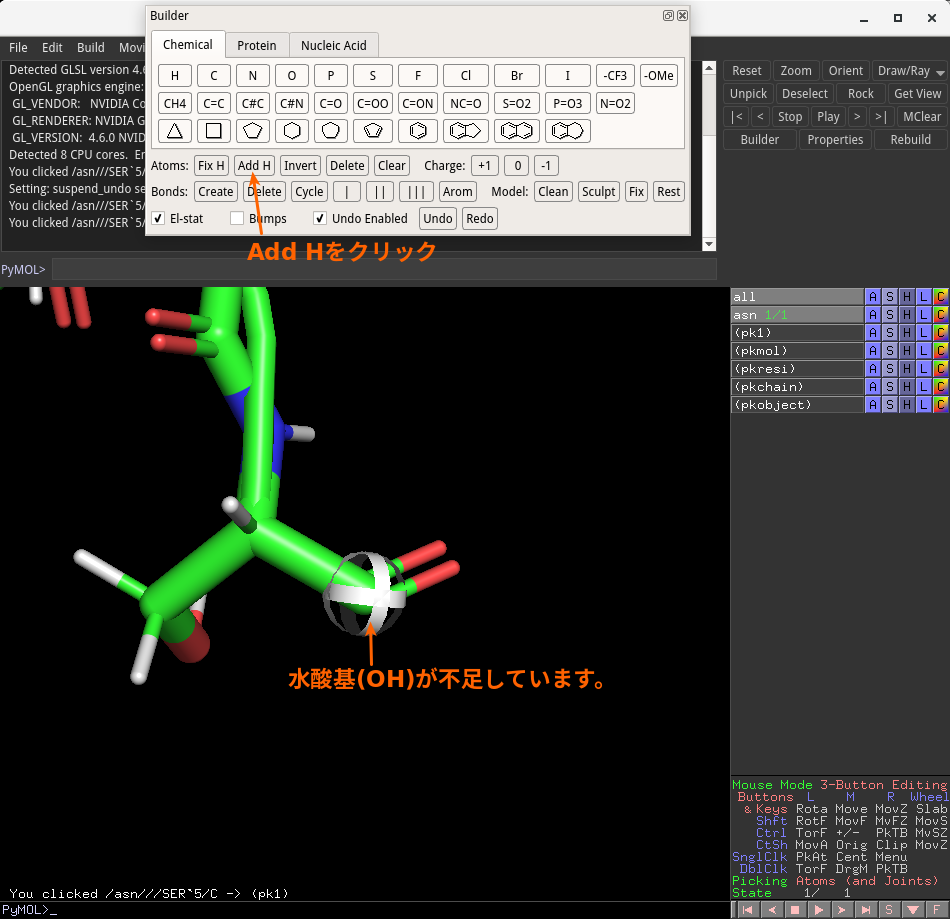
上のように、カルボニル炭素が選択された状態となっていると思いますので、その状態でBuilderウインドウの「Add H」をクリックします。
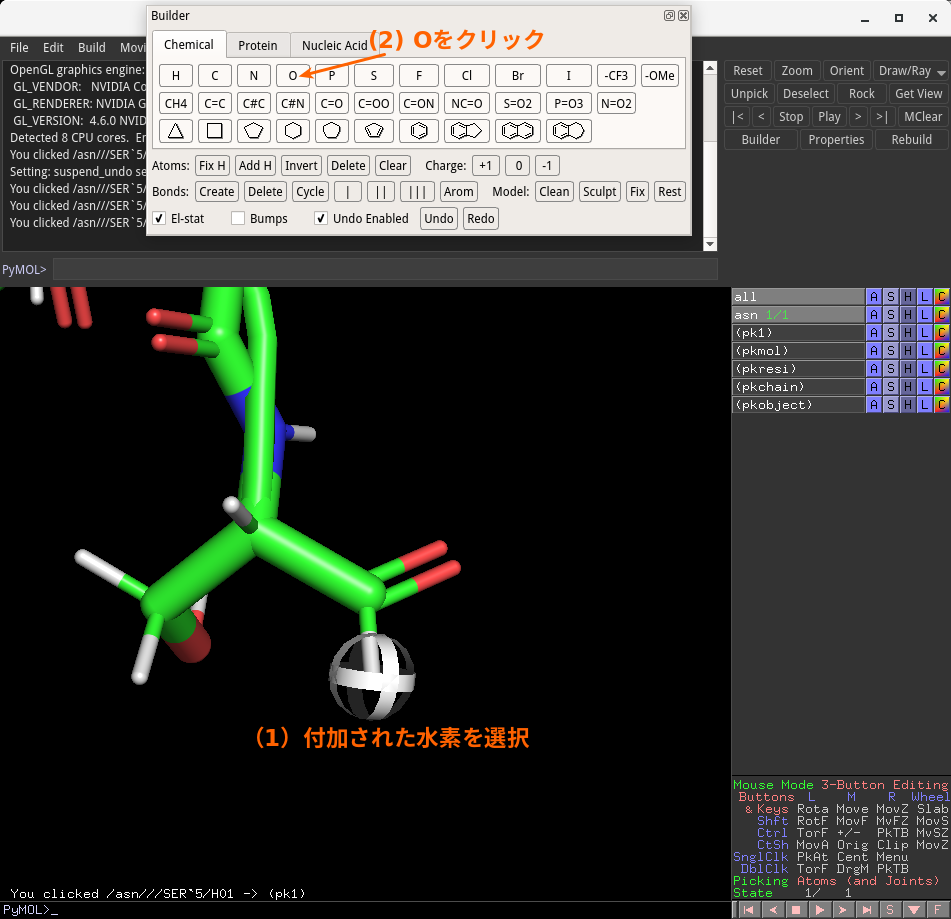
水素原子が付加された構造が得られました。次に、(1)付加された水素をクリック選択し、(2)Builderウインドウの「O」をクリックします。
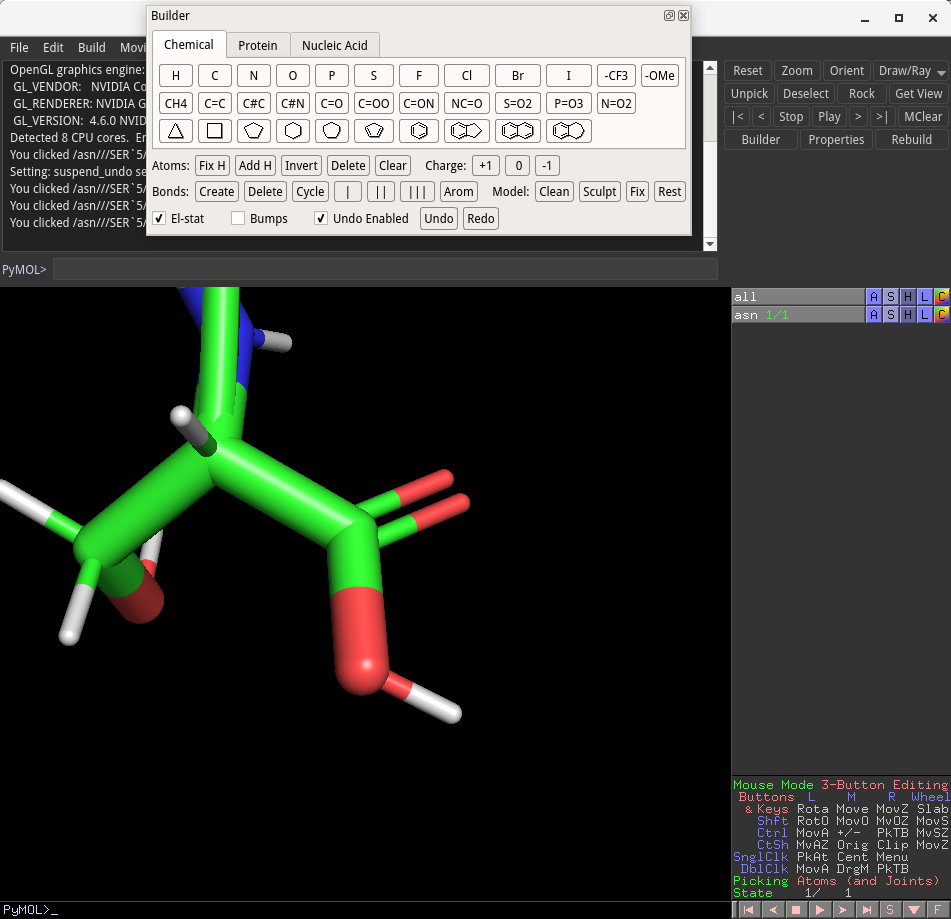
上のように、C末端に水酸基が付加できました。これでフォールディング前のTrp-cage構造のモデリングが完了しました。最後に、現時点では、全ての水素原子を削除した構造をPDBファイルとして保存したいと思います。
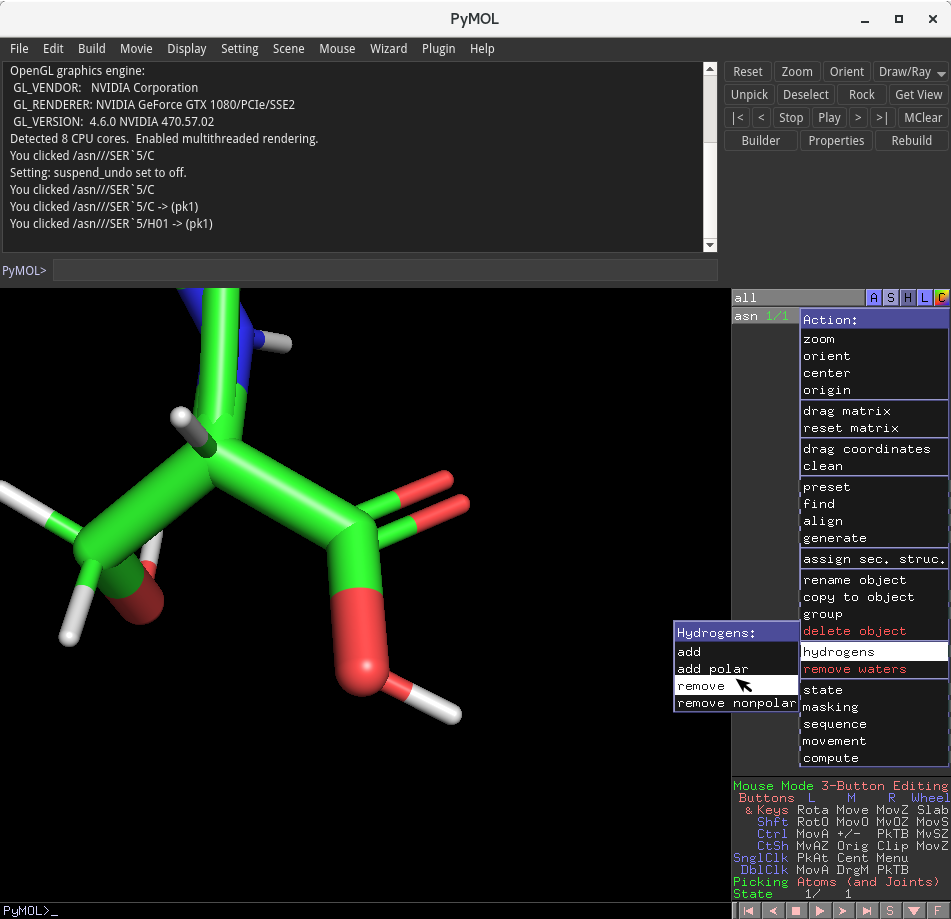
右端メニュー(asn 1/1) から、 A(Action) → hydrogens → remove を選択することで、削除できます。
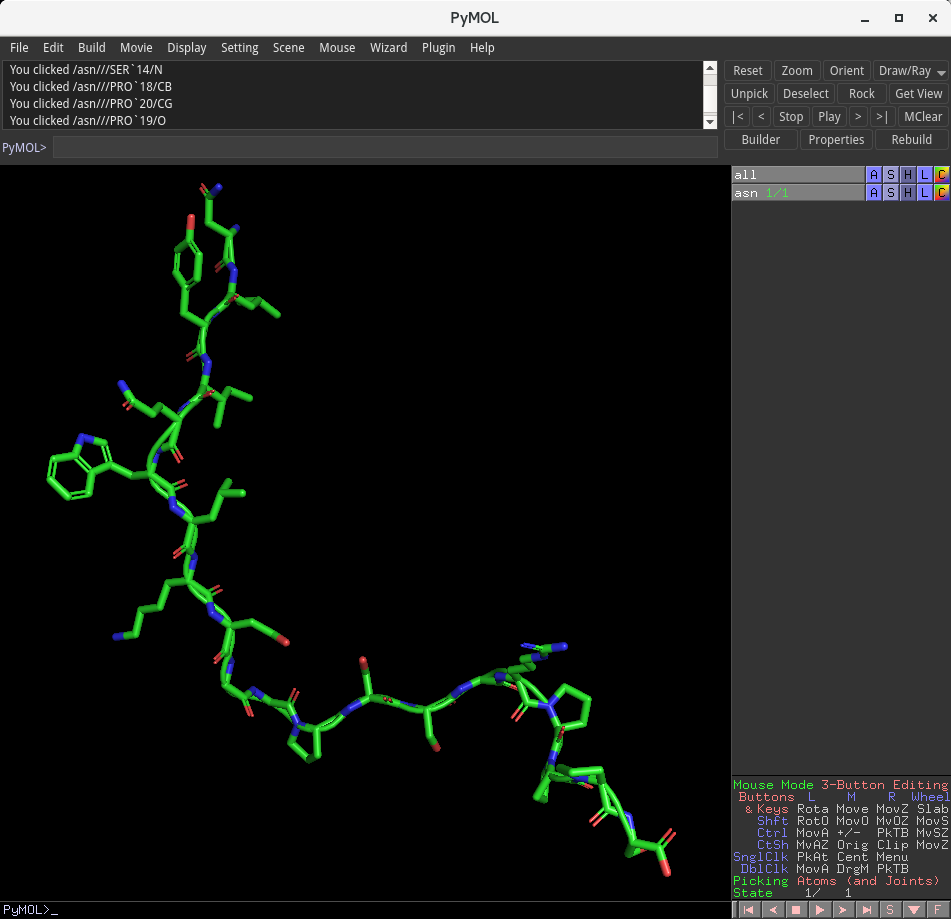
全ての水素原子が削除されました。File メニューからExport Molecule…を選択し、PDBファイルとして保存してください。ファイル名はTrpCage.pdbとしました。ファイルは、こちらのリンクからダウンロード可能です。
次回以降、このTrp-cage の構造(フォールディング前の伸びた構造)を初期構造として、OpenMMにてタンパク質フォールディングをシミュレーションしてみたいと思います。以下の記事をご覧ください。


コメント