このカテゴリーの記事では、「Pythonを使った機械学習やケモ・バイオインフォマティクスの実装や論文」を紹介していきたいと考えています。Python は3系(3.7)、anacondaを中心にして環境構築していきます。以下のようなハード・ソフト環境(CentOS Linux)を用いますが、Python(anaconda)が利用できれば、異なる環境下でも類似の実装が可能かと思います。
- CPU Intel® Core™ i7-6700K CPU @ 4.00GHz × 8
- メモリ 64GB
- GPU GeForce GTX 1080/PCIe/SSE2
- OS CentOS Linux 8
- Python 3.7
はじめに
condaによる BLAST のインストール
タンパク質の一次構造や、その遺伝子のDNA塩基配列に明らかな類似性がある場合、それらが共通の祖先から変異・進化した産物であることが推定できます。そのようなタンパク質は互いに相同性がある、相同であるといいます。類似性がある場合でも、必ずしも相同であるとはかぎらない(共通の祖先由来ではないこともある)わけですが、一次構造の類似性に基づいて、データベースからタンパク質を検索することを、一般に「相同性検索」と呼びます。
この記事では、相同性検索に汎用されるソフトフェア、「blast」をPythonのパッケージとしてインストールし、Local (ローカル) 環境での簡単な使い方を紹介します。 以前の記事と同様に、仮想環境 blast をつくり、そこにインストールしていきます。仮想環境やパッケージについては、こちらの記事を参考にして下さい。まずはターミナルで、
$ conda create -n blast python=3.7
を実行します。-n の後ろは、作成する仮想環境の名前です。今回は blast としました。 この環境 blast をアクティブにしましょう。
$ conda activate blast
を実行してください。この環境に blastパッケージをインストールしましょう。次のコマンドを実行してください。
$ conda install -c bioconda blast
依存関係にある多くのパッケージが同時にインストールされますが、y を選択してインストールを完了してください。conda list を実行すれば、blast がインストールされたのが確認できます。
タンパク質の一次構造データのダウンロード
今回の記事では、コンピュータ上にあらかじめデータベースを用意し、ローカルでのblast 検索を行ってみます。データベースの用意が必要になりますが、独自の検索が可能であったり、多数の検索を一度に実行・処理することも可能です。例として、ヒトの全てのタンパク質をデータベースにしてみます。そのために、ヒトの全てのタンパク質の一次構造データをダウンロードします。NCBI*のリンク (https://www.ncbi.nlm.nih.gov/genome/?term=Human )を参照します。これはヒトの全ての遺伝情報、ヒトゲノムの情報のまとめページですが、このページ中の protein と記されたリンクから、ヒトのタンパク質全て(推定)の一次構造を fasta という形式でダウンロードできます。
*NCBI、アメリカ国立生物工学情報センター(National Center for Biotechnology Information)は、アメリカ合衆国の国立衛生研究所 (NIH) の下の国立医学図書館 (National Library of Medicine; NLM) の一部門として 1988年11月4日に設立された機関。バイオテクノロジーや分子生物学に関連する一連のデータベースの構築及び運営、そして研究に用いられるソフトウェアの開発を行っており、バイオインフォマティクスにおける重要なリソースとなっている(ウィキペディアより)。
ここでは、以下のようにホームディレクトリに huma$ conda create -n blast python=3.7n_protein というディレクトリを作成し、wget コマンドでリンクからダウンロードしてみます(リンクが変更される場合がありますので、その際には、NCBI のホームページを参照してください。)。
$ mkdir human_protein
$ cd human_protein
$ wget https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/001/405/GCF_000001405.39_GRCh38.p13/GCF_000001405.39_GRCh38.p13_protein.faa.gz
human_protein ディレクトリに、GCF_000001405.39_GRCh38.p13_protein.faa.gzというファイル名で保存されますので、これを以下のように解凍してください。
$ gzip -d GCF_000001405.39_GRCh38.p13_protein.faa.gz
解凍して出来たファイルをテキストエディタで開いてみてください。ヒトの全てのタンパク質(推定を含む)の一次構造が並べられています。fasta 形式の詳細については、こちらを参照してください。
一次構造のデータベース化
先ほどダウンロード・解凍した fasta ファイルを指定する形で、下記のように makeblastdb コマンドを実行します。これは、blast をインストールした際に同時に使えるようになっているコマンドですので、仮想環境blastをアクティブにした状態で、実行してください。
$ makeblastdb -in GCF_000001405.39_GRCh38.p13_protein.faa -dbtype prot
上記の-in の後ろに fasta 形式の一次構造データを指定しますので、先程解凍したファイル名を指定しています。また、-dbtype prot の部分は、今回の配列がタンパク質のアミノ酸配列データであることを指定しています。
Building a new DB, current time: 03/14/2021 16:35:38 New DB name: /home/xxxx/human_protein/GCF_000001405.39_GRCh38.p13_protein.faa New DB title: GCF_000001405.39_GRCh38.p13_protein.faa Sequence type: Protein Keep MBits: T Maximum file size: 1000000000B Adding sequences from FASTA; added 116149 sequences in 2.39446 seconds.
上のような表示が出て、〜.faa.phr、〜.faa.pin、〜.faa.psq という3つのファイルが作成されているかと思います。これでデータベースが完成しました。
BLASTの使い方
ここまでの手順によって、ヒトの全タンパク質に対しての相同性検索を実施できるようになりました。実際にどのような結果が得られるのか、簡単な例を試して今回の記事を終えたいと思います。ハツカネズミ(マウス)の ミオグロビン の一次構造を使って、上で作成したヒトの全タンパク質のデータベースに対して相同性検索を行ってみます。マウスの ミオグロビン の一次構造は、UniProt というデータベースのこちらから取得できます。これをテキストファイルとして保存します。ここでは myg.fasta という名前にしました。あとは、以下のコマンドでBLAST検索が実行できます。
$ blastp -query myg.fasta -db GCF_000001405.39_GRCh38.p13_protein.faa -out myg.out
検索の問合せに用いる配列を query (クエリ)と呼びます。今回の例では、myg.fasta で保存したマウスのミオグロビンが query で、-query の後ろにファイル名を指定しています。また、-out の後ろに、検索結果の保存ファイル名を指定しています。
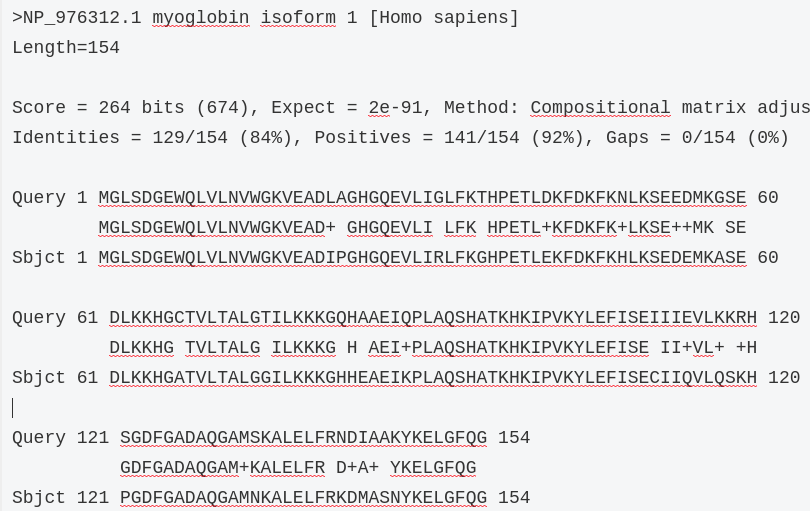
myg.out ファイルには、以下のような結果が書き込まれていると思います。
BLASTP 2.9.0+ Reference: Stephen F. Altschul, Thomas L. Madden, Alejandro A. Schaffer, Jinghui Zhang, Zheng Zhang, Webb Miller, and David J. Lipman (1997), "Gapped BLAST and PSI-BLAST: a new generation of protein database search programs", Nucleic Acids Res. 25:3389-3402. Reference for composition-based statistics: Alejandro A. Schaffer, L. Aravind, Thomas L. Madden, Sergei Shavirin, John L. Spouge, Yuri I. Wolf, Eugene V. Koonin, and Stephen F. Altschul (2001), "Improving the accuracy of PSI-BLAST protein database searches with composition-based statistics and other refinements", Nucleic Acids Res. 29:2994-3005. Database: GCF_000001405.39_GRCh38.p13_protein.faa 116,149 sequences; 77,159,190 total letters Query= sp|P04247|MYG_MOUSE Myoglobin OS=Mus musculus OX=10090 GN=Mb PE=1 SV=3 Length=154 Score E Sequences producing significant alignments: (Bits) Value NP_976312.1 myoglobin isoform 1 [Homo sapiens] 264 2e-91 ・ ・ ・ >NP_976312.1 myoglobin isoform 1 [Homo sapiens] Length=154 Score = 264 bits (674), Expect = 2e-91, Method: Compositional matrix adjust. Identities = 129/154 (84%), Positives = 141/154 (92%), Gaps = 0/154 (0%) Query 1 MGLSDGEWQLVLNVWGKVEADLAGHGQEVLIGLFKTHPETLDKFDKFKNLKSEEDMKGSE 60 MGLSDGEWQLVLNVWGKVEAD+ GHGQEVLI LFK HPETL+KFDKFK+LKSE++MK SE Sbjct 1 MGLSDGEWQLVLNVWGKVEADIPGHGQEVLIRLFKGHPETLEKFDKFKHLKSEDEMKASE 60 Query 61 DLKKHGCTVLTALGTILKKKGQHAAEIQPLAQSHATKHKIPVKYLEFISEIIIEVLKKRH 120 DLKKHG TVLTALG ILKKKG H AEI+PLAQSHATKHKIPVKYLEFISE II+VL+ +H Sbjct 61 DLKKHGATVLTALGGILKKKGHHEAEIKPLAQSHATKHKIPVKYLEFISECIIQVLQSKH 120 Query 121 SGDFGADAQGAMSKALELFRNDIAAKYKELGFQG 154 GDFGADAQGAM+KALELFR D+A+ YKELGFQG Sbjct 121 PGDFGADAQGAMNKALELFRKDMASNYKELGFQG 154

検索によって、ヒトの ミオグロビン( myoglobin isoform 1 [Homo sapiens] )がトップヒットしていますね。
Queryと記されている行がマウスのミオグロビン、Sbjctと記されている行が検索で抽出されたヒトのミオグロビン、それぞれのアミノ酸配列を表しています。また、Identities = 129/154 (84%)と記されており、両者は84%のアミノ酸が一致していることが示されています。
今回構築した環境を用いて、次回さらにBLAST検索を使った解析を進めてみたいと思います。



コメント