このカテゴリーの記事では、「Pythonを使った機械学習やケモ・バイオインフォマティクスの実装や論文」を紹介していきたいと考えています。Python は3系(3.7)、anacondaを中心にして環境構築していきます。以下のようなハード・ソフト環境(CentOS Linux)を用いますが、Python(anaconda)が利用できれば、異なる環境下でも類似の実装が可能かと思います。
- CPU Intel® Core™ i7-6700K CPU @ 4.00GHz × 8
- メモリ 64GB
- GPU GeForce GTX 1080/PCIe/SSE2
- OS CentOS Linux 8
- Python 3.7
condaによる PyMOL のインストール
この記事では、タンパク質の立体構造をパソコン上で表示したり、分析するために汎用されるソフトフェア、 “PyMOL” をインストールし、使えるようにします。前回記したように、conda create と conda install を利用して、環境を構築することができます。
まずはターミナルで、
$ conda create -n pymol python=3.7
を実行します。-n の後ろは、作成する仮想環境の名前となります。自由に変更することができますが、今回は pymol としました。
ターミナルでconda info -e を実行してみてください。
(base) [xxxx]$ conda info -e
# conda environments:
#
/home/xxxx/.local/share/r-miniconda
/home/xxxx/.local/share/r-miniconda/envs/r-reticulate
base * /home/xxxx/anaconda3
pymol /home/xxxx/anaconda3/envs/pymol
/home/xxxx/anaconda3/envs/pymol というディレクトリが作成され、環境 pymol が出来ています。この環境 pymol をアクティブにしましょう。
$ conda activate pymol
を実行してください。環境 pymol に入りました。
この環境に PyMOL をインストールしましょう。次のコマンドを実行してください。
$ conda install -c schrodinger pymol
「PyMOLはオープンソースの分子グラフィックスツールである。ウォーレン・デラノにより開発され、個人経営のソフトフェア会社であるデラノ・サイエンティフィック によって商品化された。現在はSchrödinger社によって商品化されている。」(ウィキペディア)ということで、上記の -c schrodinger という引数を使うことでインストールできます(*この状態では、PyMOLの機能のいくつかが制限されています。Schrödinger社のライセンスを導入すれば、機能制限を解くこともできますが、このままでも問題なく使うことができます。)。依存関係にある多くのパッケージが同時にインストールされますが、y を選択してインストールを完了してください。conda list を実行すれば、pymol 2.4.1 がインストールされたのが確認できると思います。
PyMOL の起動
(*今回、環境 pymol を用意して、そのなかでパッケージ pymol をインストールしました。そのため、 パッケージ pymol を実行して、ソフトウェアを起動する際、環境 pymol がアクティブになっている( conda activate pymol した状態)必要があります。環境を用意せずに、base 環境に直接パッケージをインストールすることも可能ですが、あえて仮想環境を用意しています。これは一見面倒ですが、異なる仮想環境を用意することで、依存関係の異なる様々なパッケージを導入することが可能になります。また、後々、使用する必要のなくなった仮想環境を削除しても、他の環境・他のパッケージに影響が及びません。)
環境 pymol をアクティブにした状態で、
$ pymol
を実行することで、PyMOLが起動されます。以下のようなウィンドウが出ますが、
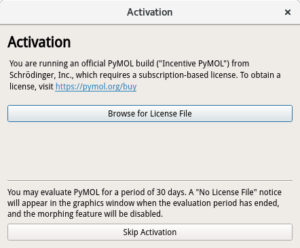
Skip Activation をクリックしてください。
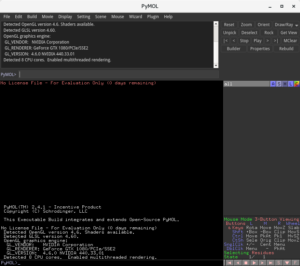
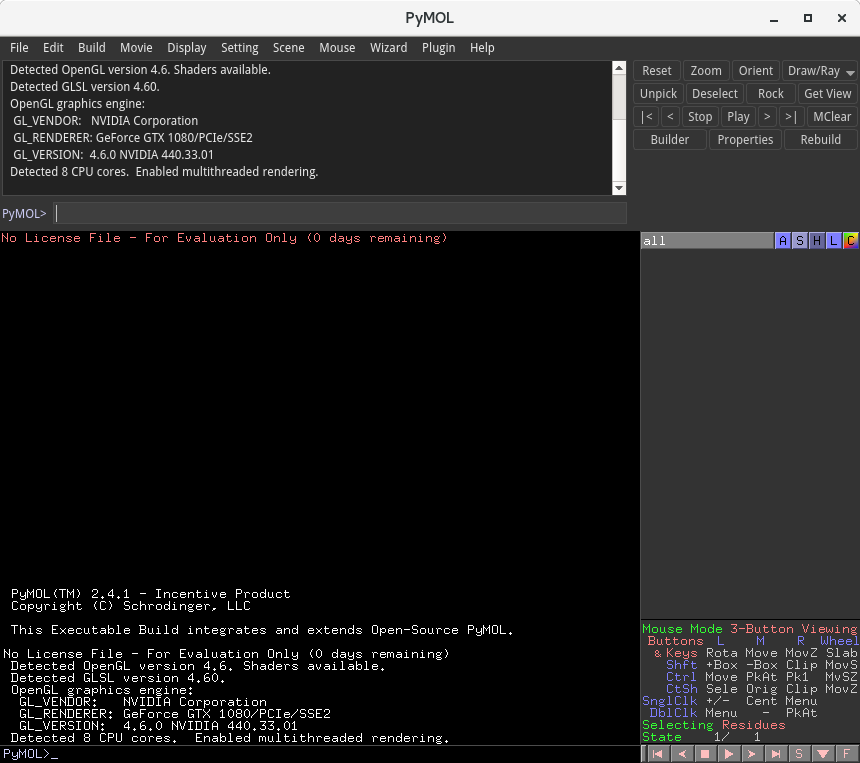
このようにPyMOLが起動します。

コメント