前回、サンプル間の実際の距離をできるだけ再現する、計量MDS(PCoA)を解説しました。

しかし、生態学の生物データやアンケート結果など、データによってはAとBの距離は2.5という正確な距離の値よりも、AとBは、AとCよりも近い、という“順序”の方が重要な場合があります。
今回は、距離の大小関係(順序)を重視して関係性を可視化する、もう一つのMDSである非計量MDS(NMDS)について、Rでの実践方法を解説します。
1. NMDS(非計量多次元尺度構成法)とは
2. 使用パッケージとデータ
3. RでNMDSを実行する
4. 結果の可視化と解釈
4-1. ステップ①:サンプル(地点)のみをプロット
4-2. ステップ②:サンプルと種を追加してプロット
4-3. ステップ③:環境変数を追加してプロット
5. ggplot2でカスタマイズしやすく
6. まとめ
1. NMDS(非計量多次元尺度構成法)とは
NMDS(Non-metric Multidimensional Scaling)は、たくさんの変数を持つデータ間の似ている/似ていないという関係性(非類似度)を、2次元や3次元のマップとして可視化する手法です。
例えば、複数の調査地点で何十種類もの生物の個体数を記録したデータは、一見、どの地点とどの地点の生物コミュニティが似ているのかを把握するのは難しいです。
NMDSは、このようなデータからサンプル間の非類似度(距離)の大小関係(順序)を計算し、その大小関係をできるだけ保ったまま、2次元や3次元の散布図にサンプルを配置し直します。
今回は、NMDSを用いて、以下の3つのステップでデータの関係性を段階的に調べていきます。
ステップ① 調査地点(サンプル)が互いにどの程度似ているのか、その全体像を可視化します。
ステップ② その地点の配置に影響を与えている生物種(説明変数)を明らかにします。
ステップ③ 地点のグループ分けの背景にある環境要因を探るため、環境変数が配置に与える影響を評価します。
2. 使用パッケージとデータ
生態学の群集データ解析で広く使われているveganパッケージを使用します。
データは、veganパッケージのvarespecとvarechemを使います。
varespecとvarechemは、トナカイを放牧する松林における地衣類(※)やコケ植物などの下層植物と土壌特性に関するデータです。varespecは24行44列で構成され、列は44種の下層植物の推定被覆率値の記録です。varechemは24行14列で、varespecと同一地点の14種類の土壌特性(環境変数)の記録です。
※地衣類:地衣類(ちいるい)は、菌類のうち、藻類を共生させることで自活できるようになった生物である(Wikipedia 地衣類 より)
土壌特性は、N:窒素, P:リン, K:カリウム, Ca:カルシウム, Mg:マグネシウム, S:硫黄, Al:アルミニウム, Fe:鉄, Mn:マンガン, Zn:亜鉛, Mo:モリブデン, Baresoil:裸地の推定被覆率, Humdepth:腐植土の厚さ, pHの数値を示します。
このデータを使ってNMDSを行います。
# veganパッケージがインストールされていない場合はインストール
install.packages("vegan")
library(vegan)
# データセットを読み込む
data(varespec)
data(varechem)
# varespecデータ(6行×6列)を確認(行が地点、列が地衣類名)
head(varespec[, 1:6])
# 出力結果 Callvulg Empenigr Rhodtome Vaccmyrt Vaccviti Pinusylv 18 0.55 11.13 0.00 0.00 17.80 0.07 15 0.67 0.17 0.00 0.35 12.13 0.12 24 0.10 1.55 0.00 0.00 13.47 0.25 27 0.00 15.13 2.42 5.92 15.97 0.00 23 0.00 12.68 0.00 0.00 23.73 0.03 19 0.00 8.92 0.00 2.42 10.28 0.12
# varechemデータ head(varechem)
# 出力結果
N P K Ca Mg S Al Fe Mn Zn Mo Baresoil Humdepth pH
18 19.8 42.1 139.9 519.4 90.0 32.3 39.0 40.9 58.1 4.5 0.3 43.9 2.2 2.7
15 13.4 39.1 167.3 356.7 70.7 35.2 88.1 39.0 52.4 5.4 0.3 23.6 2.2 2.8
24 20.2 67.7 207.1 973.3 209.1 58.1 138.0 35.4 32.1 16.8 0.8 21.2 2.0 3.0
27 20.6 60.8 233.7 834.0 127.2 40.7 15.4 4.4 132.0 10.7 0.2 18.7 2.9 2.8
23 23.8 54.5 180.6 777.0 125.8 39.5 24.2 3.0 50.1 6.6 0.3 46.0 3.0 2.7
19 22.8 40.9 171.4 691.8 151.4 40.8 104.8 17.6 43.6 9.1 0.4 40.5 3.8 2.7
3. RでNMDSを実行する
veganパッケージのmetaMDS()関数を使ってNMDSを実行します。NMDSは、ランダムな初期配置から計算を始めるため、set.seed()で乱数を固定しておきます。
# 乱数を固定して結果を再現可能にする set.seed(123) # varespec(種構成データ)のみを使ってNMDSを実行 # 距離はブレイ・カーティス非類似度(デフォルト) nmds_result <- metaMDS(varespec)
# 実行中の出力例 Square root transformation Wisconsin double standardization Run 0 stress 0.1843196 Run 1 stress 0.2245479 Run 2 stress 0.215148 Run 3 stress 0.2324692 Run 4 stress 0.1985581 Run 5 stress 0.1843196 ... New best solution ... Procrustes: rmse 3.634903e-05 max resid 0.0001465453 ... Similar to previous best Run 6 stress 0.1974408 Run 7 stress 0.3840757 Run 8 stress 0.2234787 Run 9 stress 0.1843196 ... New best solution ... Procrustes: rmse 1.889957e-05 max resid 7.643299e-05 ... Similar to previous best Run 10 stress 0.1852397 Run 11 stress 0.2090733 Run 12 stress 0.2066172 Run 13 stress 0.1982376 Run 14 stress 0.196245 Run 15 stress 0.18584 Run 16 stress 0.2085949 Run 17 stress 0.1985582 Run 18 stress 0.1985581 Run 19 stress 0.2079056 Run 20 stress 0.2126569 *** Best solution repeated 1 times
metaMDS()は、最適な解を見つけるために異なる初期値から計算を繰り返します(今回はRun0~20の21回。デフォルトで20回以上)。
# 結果の概要を表示
nmds_result
# 出力結果
Call:
metaMDS(comm = varespec)
global Multidimensional Scaling using monoMDS
Data: wisconsin(sqrt(varespec))
Distance: bray
Dimensions: 2 #1
Stress: 0.1843196 #2
Stress type 1, weak ties
Best solution was repeated 1 time in 20 tries #3
The best solution was from try 9 (random start)
Scaling: centring, PC rotation, halfchange scaling
Species: expanded scores based on ‘wisconsin(sqrt(varespec))’
#1 結果は2次元マップとして作成されました。
#2 ストレス値は0.184で、0.2を下回るため、解釈に使える良いモデルです(※)。
#3 21回の試行の中で、最も低いStress値を持つ安定した解が1種類見つかりました。
※💡ストレス値とは?
NMDSでは、元のデータの距離の大小関係を2次元あるいは3次元のマップでどれだけ忠実に再現できたか、その当てはまりの悪さを示す指標としてストレス値が計算されます。
この値は当てはまりの良さを示す数値として用いられ、0に近いほど良いモデルです。以下のような目安で判断されます※。
0.025: 素晴らしい
0.05: よい
0.1: まずまず
0.2: 悪い
※参考図書 金 明哲, Rによるデータサイエンス
※参考文献 土居 秀幸, 岡村 寛, 生物群集解析のための類似度とその応用 : Rを使った類似度の算出、グラフ化、検定, 日本生態学会誌, 2011, 61 巻, 1 号, p. 3-20, https://doi.org/10.18960/seitai.61.1_3,
今回のストレス値は0.2を下回っており、信頼できる結果だと判断できます。
4. 結果の可視化と解釈
NMDSの結果を、情報を追加しながら上記の①~③のステップで可視化していきます。
4-1. ステップ①:サンプル(地点)のみをプロット
サンプル(地点)の関係性だけを見て、全体像を掴みます。
# 地点のみをプロット(テキストラベルで表示) plot(nmds_result, display = "sites", type = "text") # nmdsプロットをサンプル地点の番号としてを記載
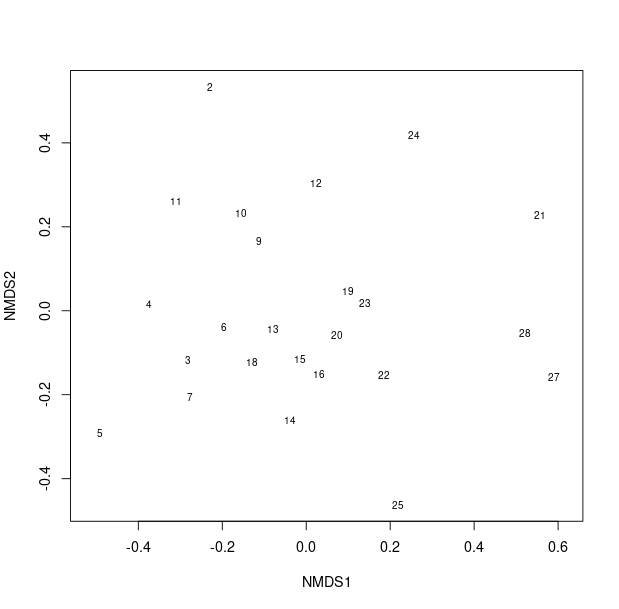
このマップ上で距離が近い地点どうしは、下層植物の構成が似ていることを意味します。例えば、中央付近の地点19,23は互いに似ていると考えられます。
4-2. ステップ②:サンプルと種を追加してプロット
次に、地点の配置にどの種が影響しているのかを見るため、種の情報も追加します。
# 地点(黒)と種(赤)を両方プロット
plot(nmds_result, display = c("sites", "species"), type = "text")
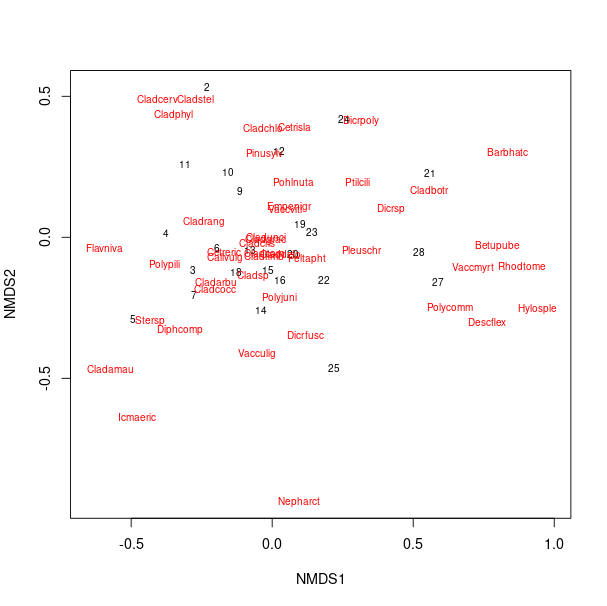
地点の近くにある種は、その地点の植生を特徴づける種であると解釈できます。例えば、左上の地点2の近くにはCladstelがあり、これらの種がこのグループの植生に強く関連していることが考えられます。
4-3. ステップ③:環境変数を追加してプロット
最後に、地点のグループ分けの背後にある理由を探るため、環境変数を矢印で重ね合わせます。envfit()関数を使います。
# envfit()で環境変数とNMDS配置の相関を計算
set.seed(123) # 結果の再現性を確保
env_fit_result <- envfit(nmds_result ~ ., data = varechem)
env_fit_result
# 出力結果
***VECTORS
NMDS1 NMDS2 r2 Pr(>r)
N -0.05039 -0.99873 0.2081 0.073 .
P 0.68702 0.72664 0.1755 0.135
K 0.82729 0.56178 0.1657 0.158
Ca 0.75015 0.66127 0.2811 0.037 *
Mg 0.69675 0.71731 0.3494 0.013 *
S 0.27626 0.96108 0.1774 0.133
Al -0.83776 0.54603 0.5155 0.001 ***
Fe -0.86197 0.50695 0.4001 0.007 **
Mn 0.80233 -0.59688 0.5322 0.002 **
Zn 0.66518 0.74668 0.1779 0.128
Mo -0.84876 0.52878 0.0517 0.569
Baresoil 0.87210 -0.48933 0.2494 0.051 .
Humdepth 0.92636 -0.37664 0.5590 0.001 ***
pH -0.79908 0.60123 0.2624 0.044 *
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Permutation: free
Number of permutations: 999
NMDS1, NMDS2: プロット上の矢印の向きを決定する座標。(矢印の向き)
r2 (決定係数): NMDSのプロットとの相関の強さを示す。0から1までの値をとり、1に近いほど相関が強い。(矢印の長さ)
Pr(>r) (p値): 相関の統計的な信頼性を示す。この値が小さいほど意味のある変数であると考えられる。
地点と種のプロットに、環境変数の矢印を重ねていきます。
p値の有意水準を0.05とし、0.05以下の矢印を表示させます。
※p.maxを使用しているので、p値0.05以下として計算しています。有意水準未満とする場合は、このあたりはggplot2の方が柔軟にカスタマイズできます。
# プロットを描画
# 地点(黒)と種(赤)のマップを作成
plot(nmds_result, display = c("sites", "species"), type = "text")
# 環境変数の矢印(青)を重ね描き
plot(env_fit_result, col = "blue", p.max=0.05)
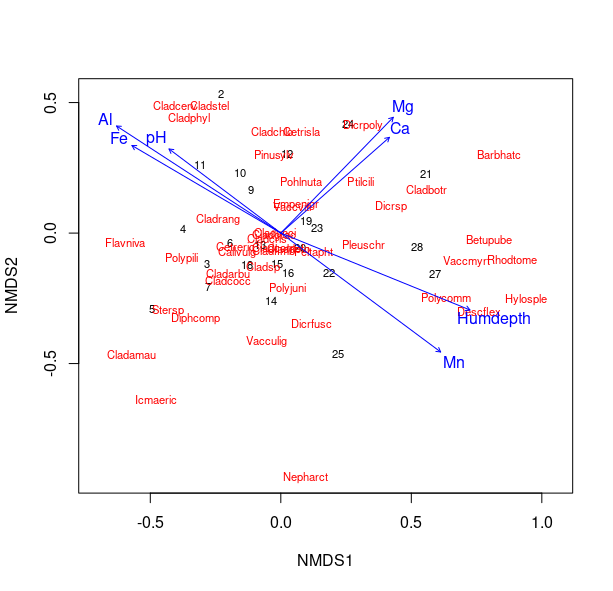
青い矢印は、サンプル(地点)の配置と強く相関する環境要因を示しています。また、これらの要因が、それらの地点を構成する植物種の分布にも影響を与える上で重要であることを示唆しています。
・矢印の方向: その環境変数の値が最も高くなる方向を示します。例えば、pHの矢印が指す方向にある地点ほど、土壌のpHが高い傾向にあります。(11はpH最も高い)
・矢印の長さ: その環境変数が、植物の分布に与える影響の強さを表します。Humdepth(腐植土の深さ)のような長い矢印は、短い矢印よりも植物群集との相関が強いことを意味します。
5. ggplot2で図をカスタマイズしやすく
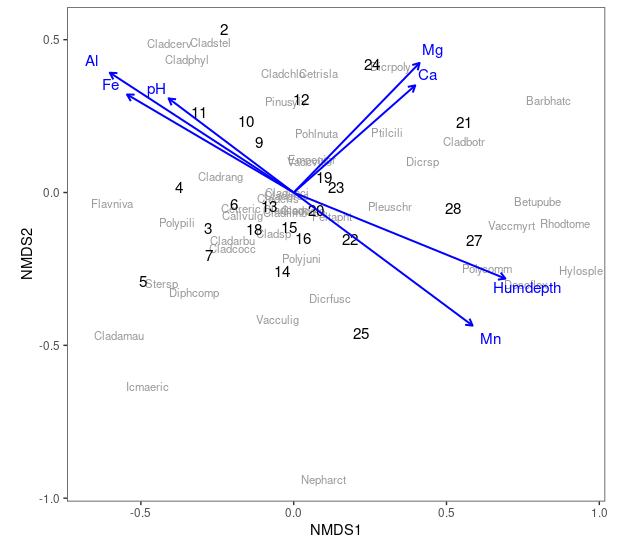
# 地点の座標を抽出 site_scores <- as.data.frame(scores(nmds_result, display = "sites")) site_scores$site_name <- rownames(site_scores) # 種の座標を抽出 species_scores <- as.data.frame(scores(nmds_result, display = "species")) species_scores$species_name <- rownames(species_scores) # 環境変数の座標を抽出 vector_scores <- as.data.frame(scores(env_fit_result, display = "vectors")) vector_scores$variable <- rownames(vector_scores) # vector_scoresにp値の列を追加 vector_scores$p_value <- env_fit_result$vectors$pvals # p値が0.05未満の行だけを抽出して、新しいデータフレームを作成 sig_vector_scores <- vector_scores[vector_scores$p_value < 0.05, ] library(ggplot2) ggplot() + # グレーで種をプロット geom_text(data = species_scores, aes(x = NMDS1, y = NMDS2, label = species_name), color = "gray60", size = 3) + # 地点(サンプル)を黒文字でプロット geom_text(data = site_scores, aes(x = NMDS1, y = NMDS2, label = site_name), color = "black", size = 4) + # p<0.05の環境変数の矢印 geom_segment(data = sig_vector_scores, aes(x = 0, y = 0, xend = NMDS1, yend = NMDS2), arrow = arrow(length = unit(0.2, "cm")), # 矢印の先の矢じりをかき、大きさを指定 color = "blue", linewidth = 0.7) + # 矢印の色と線の幅指定 # p<0.05の環境変数のラベルをプロット(矢印の少し先に表示) geom_text(data = sig_vector_scores, aes(x = NMDS1 * 1.1, y = NMDS2 * 1.1, label = variable), color = "blue", size = 4) + theme_test() + coord_fixed() # 縦横比を1:1に固定して距離の歪みをなくす
6. まとめ
今回は、距離の「大小関係(順序)」を重視する非計量MDS(NMDS)について、段階的な可視化を通して解説しました。
キーポイント:
・NMDSは、生態学で標準的に使われるブレイ・カーティス非類似度と相性が良く、非線形なデータの複雑な構造を捉えるのに適しています。
・分析は、①サンプルの全体像の把握、②種の関連性の特定、③環境変数によるパターンの説明、というステップで進めることで、深い洞察が得られます。
・Rではveganパッケージが強力なツールとなり、metaMDS()で計算し、envfit()で環境要因を評価する流れを紹介しました。
前回の計量MDSと今回の非計量MDS、それぞれの長所とアプローチの違いを理解し、データの性質や分析の目的に合わせて使い分けてみてください。




コメント