ロジスティック回帰は、二値分類問題(例:疾病リスク予測、顧客解約予測)に広く用いられています。
先日、Rでのロジスティック回帰分析について解説しました。

ステップワイズ法は、AICなどで変数を追加・削除しながら最適なモデルを選択する方法です。この方法では、変数が多いと学習データに過度に適合(過学習, オーバーフィッティング)しやすく、テストデータに対する予測精度が低下する可能性があります。その結果、汎化性の低いモデルとなり、未知のデータに対する予測が不安定になります。また、相関の強い変数(多重共線性)が存在すると、不適切な変数が選ばれるリスクがあり、解釈が難しくなることがあります。多重共線性のあるデータでは、ステップワイズ法の使用は慎重に検討すべきです。
過学習の防止や、多重共線性の影響を考慮する方法として、LASSO回帰やRidge回帰などの”正則化“を用いる方法があります。
正則化とは、過学習を防ぐためにモデルの複雑さを抑える手法 です。具体的には、損失関数(予測値と実測値の誤差を表す関数)に正則化項を追加し、回帰係数の大きさを制限 することで、シンプルで汎化性能の高いモデルを作成します。代表的な手法として、LASSO回帰(L1正則化) や Ridge回帰(L2正則化) があります。
今回は、”正則化“を用いたロジスティック回帰分析について解説していきます。
1. 正則化ロジスティック回帰モデルの基礎
2. データの準備と前処理
3. LASSO回帰によるロジスティック回帰モデル(L1正則化)
4. Ridge回帰によるロジスティック回帰モデル(L2正則化)
5. LASSO・Ridge回帰を適用したロジスティック回帰モデルの比較
1. 正則化ロジスティック回帰モデルの基礎
◆正則化項の種類
・LASSO回帰(L1正則化): 回帰係数の絶対値の和をペナルティ項として加える。一部の回帰係数を0にする効果があり、重要な特徴だけを残したい場合の変数選択に有効。
・ Ridge回帰(L2正則化): 回帰係数の二乗和をペナルティ項として加える。全ての変数の係数を縮小し、多重共線性の問題を緩和する。
・Elastic Net: L1正則化とL2正則化を組み合わせた手法で、両方のメリットを活かせる。
◆ハイパーパラメータ
・正則化の強さを決めるハイパーパラメータ(λなど)の調整が重要。
・ハイパーパラメータの値が大きすぎると、モデルが単純になりすぎて逆に性能が低下する可能性がある。
・クロスバリデーションなどを使って、最適なハイパーパラメータを探索する。
◆手法特徴まとめ
| 手法 | 特徴 | メリット | デメリット | 適しているケース |
| ステップワイズ法 | 変数を1つずつ追加・削除 | 計算量が少なく、作業がシンプル | 過学習のリスク、変数の相関関係を考慮しにくい | 変数の数が比較的少なく、相関関係が低い場合 |
| LASSO回帰 | L1正則化により、不要な変数の係数を0にする | 変数選択とモデルの簡素化を同時に行える、スパースな解が得られる(※) | ハイパーパラメータ(※)の調整が必要、情報を完全に削除するリスク | 変数が多い場合、スパースな解を求めたい場合 |
| Ridge回帰 | L2正則化により、変数の係数を縮小 | 多重共線性を解消し、モデルの安定性を向上 | 変数の係数を0にすることはできない、ハイパーパラメータの調整が必要 | 多重共線性の問題がある場合 |
| Elastic Net | L1とL2正則化を組み合わせ | LASSOとRidge回帰の両方のメリットを活かせる | ハイパーパラメータを2つ調整する必要がある | LASSOとRidge回帰のどちらが適切か判断できない場合 |
補足
※スパースな解:スパース(Sparse)は「まばらな」という意味。多くの変数の係数をゼロとして、モデルをより単純化した解のことをさす。
※ハイパーパラメータ:モデルの学習前に手動で設定する制御パラメータ であり、学習率や正則化強度(λ)などが含まれる。
2. データの準備と前処理
使用するデータセットは、Pima.trで、21歳以上の女性を対象に世界保健機関の基準に従って糖尿病の検査データです。
データセットの変数
npreg:妊娠回数、glu:血糖値、bp:血圧、skin:皮膚の厚さ、bmi:BMI、ped:糖尿病の家族歴(遺伝的リスク)、age:年齢、type:糖尿病の有無(Yes or No)
このデータを用いて、糖尿病の有無(二値分類)を予測するロジスティック回帰モデルを作成します。
ロジスティック回帰モデル作成のために、以下の処理を行います:
・目的変数 (type) を二値変換(1 = “Yes”, 0 = “No”)
・説明変数のスケーリング(※1)
・学習データとテストデータに分割(70%を学習データとする)
※1 説明変数のスケーリングは、特に正則化を使用する場合必要です。
正則化手法(LASSO, Ridge, Elastic Net)は、回帰係数の大きさにペナルティを課すため、変数のスケールが異なると影響が偏るためです。例えば、異なる単位の変数が混在すると、数値の大きい変数が優先的に影響を受けるため、適切な変数選択やモデル精度が損なわれてしまいます。
install.packages(c("glmnet", "MASS", "caret", "pROC"))
library(glmnet)
library(MASS)
library(caret)
library(pROC)
data(Pima.tr)
head(Pima.tr)
# npreg glu bp skin bmi ped age type
#1 5 86 68 28 30.2 0.364 24 No
#2 7 195 70 33 25.1 0.163 55 Yes
#3 5 77 82 41 35.8 0.156 35 No
#4 0 165 76 43 47.9 0.259 26 No
#5 0 107 60 25 26.4 0.133 23 No
#6 5 97 76 27 35.6 0.378 52 Yes
##目的変数 (type) を二値変換(1 = "Yes", 0 = "No")
Pima.tr$type<-ifelse(Pima.tr$type=="Yes",1,0)
x <- as.matrix(Pima.tr[, -ncol(Pima.tr)]) # 説明変数
y <- Pima.tr$type
##説明変数のスケーリング
x_scaled <- scale(x)
##学習データとテストデータに分割
set.seed(123)
train_index <- createDataPartition(y, p = 0.7, list = FALSE) ##データの 70% を学習データとする
x_train <- x_scaled[train_index, ]
y_train <- y[train_index]
x_test <- x_scaled[-train_index, ]
y_test <- y[-train_index]
3. LASSO回帰によるロジスティック回帰モデル(L1正則化)
LASSO回帰による変数選択を行うロジスティック回帰モデルの作成手順:
・ LASSO回帰によるロジスティック回帰モデルの作成(クロスバリデーションで最適なλに決定)
・変数選択
・モデルの評価(ROC曲線、AUC)
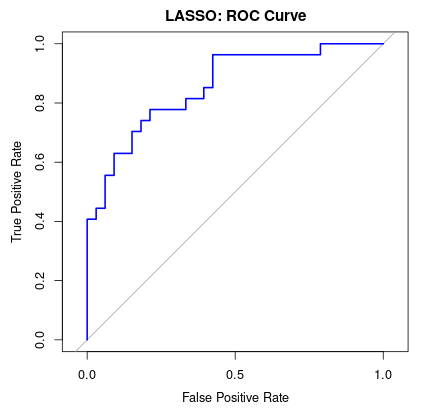
##LASSO回帰を適用したロジスティック回帰をクロスバリデーションで実行し、最適なλを選択するモデルを作成。 lasso_model <- cv.glmnet(x_train, y_train, family = "binomial", alpha = 1) ##family = "binomial"でロジスティック回帰、alpha=1でLASSO lasso_model #Call: cv.glmnet(x = x_train, y = y_train, family = "binomial", alpha = 1) #Measure: Binomial Deviance # Lambda Index Measure SE Nonzero #min 0.02450 23 0.982 0.05252 6 #1se 0.07483 11 1.034 0.04095 5 ##最適なλ(Lambda.min): 0.02450 ##LASSO回帰(L1正則化)の最適なλ(lambda.min)で推定された回帰係数の確認 lasso_coeffs <- coef(lasso_model, s = lasso_model$lambda.min) print(lasso_coeffs) #8 x 1 sparse Matrix of class "dgCMatrix" # s1 #(Intercept) -1.0192222 #npreg 0.3415534 #glu 0.6267927 #bp . #skin 0.1508677 #bmi 0.2459345 #ped 0.1876616 #age 0.3403301 ## logit(p)=−1.0192+(0.3416×npreg)+(0.6268×glu)+(0.1509×skin)+(0.2459×bmi)+(0.1877×ped)+(0.3403×age) ##ROC曲線を作成 lasso_pred <- predict(lasso_model, newx = x_test, s = lasso_model$lambda.min, type = "response") roc_curve_lasso <- roc(y_test, lasso_pred) #Setting levels: control = 0, case = 1 #Setting direction: controls < cases #Warning message: #In roc.default(y_test, lasso_pred) : # Deprecated use a matrix as predictor. Unexpected results may be produced, please pass a numeric vector. ## AUC算出 auc(roc_curve_lasso) ##作図 plot(roc_curve_lasso, col = "blue", main = "LASSO: ROC Curve", legacy.axes = TRUE,xlab = "False Positive Rate", ylab = "True Positive Rate")
LASSO回帰で変数選択し、変数bpは削除されたモデルになりました。
logit(p)=−1.0192+(0.3416×npreg)+(0.6268×glu)+(0.1509×skin)+(0.2459×bmi)+(0.1877×ped)+(0.3403×age)
AUCは、0.856であり、Very good(とてもよい)であることが分かりました(※2)。
※2 AUC数値の基準 参考文献
Trifonova OP, Lokhov PG, Archakov AI. [Metabolic profiling of human blood]. Biomed Khim. 2014 May-Jun;60(3):281-94. Russian. doi: 10.18097/pbmc20146003281. PMID: 25019391.
4. Ridge回帰によるロジスティック回帰モデル(L2正則化)
Ridge回帰によるロジスティック回帰モデルも、LASSO回帰のときと同様の手順ですすめます。
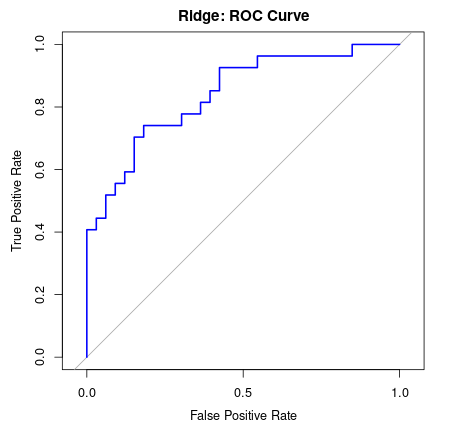
ridge_model <- cv.glmnet(x_train, y_train, family = "binomial", alpha = 0) ridge_model #Call: cv.glmnet(x = x_train, y = y_train, family = "binomial", alpha = 0) #Measure: Binomial Deviance # Lambda Index Measure SE Nonzero #min 0.0636 87 0.9618 0.09493 7 #1se 0.6508 62 1.0539 0.07121 7 ##最適なλ(Lambda.min): 0.0636 ridge_coeffs <- coef(ridge_model, s = ridge_model$lambda.min) print(ridge_coeffs) #8 x 1 sparse Matrix of class "dgCMatrix" # s1 #(Intercept) -1.01177443 #npreg 0.36086975 #glu 0.56116125 #bp -0.08105387 #skin 0.22072282 #bmi 0.26500298 #ped 0.25414735 #age 0.37870368 ridge_pred <- predict(ridge_model, newx = x_test, s = ridge_model$lambda.min, type = "response") roc_curve_ridge <- roc(y_test, ridge_pred) #Setting levels: control = 0, case = 1 #Setting direction: controls < cases #Warning message: #In roc.default(y_test, ridge_pred) : # Deprecated use a matrix as predictor. Unexpected results may be produced, please pass a numeric vector. plot(roc_curve_ridge, col = "blue", main = "Ridge: ROC Curve", legacy.axes = TRUE,xlab = "False Positive Rate", ylab = "True Positive Rate") auc(roc_curve_ridge) #Area under the curve: 0.8406
AUCは、0.8406であり、Very good(とてもよい)であることが分かりました(※2)。
5. LASSO・Ridge回帰を適用したロジスティック回帰モデルの比較
LASSO回帰とRidge回帰を適用したロジスティック回帰モデルを比較しました。
LASSO回帰では、不要な変数としてbpが削除され、スパースなモデルを構築することで、AUCは0.856と高い分類性能を示しました。
一方、Ridge回帰ではすべての変数を残しつつ係数を縮小しており、AUCは0.8406と、LASSOの場合に比べると若干低い結果でした。
LASSO回帰は不要な変数を削除し、解釈しやすいシンプルなモデルを作成できるのが強みです。今回の結果でも、LASSO回帰によってモデルの精度を維持しつつ変数を減らすことができました。ただし、すべての変数を活かしたい場合や、多重共線性が強い場合にはRidge回帰の方が適していることもあります。
二値の予測を行うロジスティック回帰モデル作成する際、過学習防止や、多重共線性の対策として、LASSOやRidge回帰などを適切に用いていただければと思います。




コメント