今回は、アップセット図(UpSet図)についてRのコード例を紹介したいと思います。

アップセット図(UpSet図)とは?
ベン図とは違うアプローチで、集合の関係性を視覚的に示す図です。
RのUpSetRパッケージについては、集合の違いを表すベン図に代わるもの、として以下の論文(※1)で紹介されています。
※1 参考文献
Jake R Conway, Alexander Lex, Nils Gehlenborg, UpSetR: an R package for the visualization of intersecting sets and their properties, Bioinformatics, Volume 33, Issue 18, September 2017, Pages 2938–2940, https://doi.org/10.1093/bioinformatics/btx364
”ベン図やオイラー図は、集合の交差を定量的に視覚化するための一般的なソリューションであるが、不十分である。交差する集合とその特性を可視化するための,ベン図やオイラー図に代わるスケーラブルなソリューションが必要である。”
ベン図の特徴:
2つまたは3つの集合を円や楕円で表し、それらの集合の重複や個別要素を示します。3つまでの集合の要素の共通性や相違点を強調する場合に一般的に使用され、集合が4つ以上に増えると理解が難しくなるともいわれています。
UpSet図の特徴:
集合の共通要素や個別要素を表す水平な棒と、それらの組み合わせを表す垂直な線で構成されます。多数の集合に対し、集合の重複や相互作用を簡潔に示したい場合に適しているとされています。
集合が多いときの共通要素を理解したい場合は、UpSet図の方が適している、というイメージですね。
ベン図のコード例で使用した遺伝子情報のダミーデータを用いて、UpSet図を作成して見ます。
↓↓↓ベン図の紹介記事です。

genes <- paste("gene",1:1000,sep="") #1
set.seed(0123)
x <- list(
A=sample(genes,100),
B=sample(genes,150),
C=sample(genes,350),
D=sample(genes,450),
E=sample(genes,200),
F=sample(genes,300),
G=sample(genes,500)) #2
install.packages("UpSetR")
library(UpSetR)
upset(fromList(x), nsets = 7) #3
#1, 2は、遺伝子情報のダミーデータ作成
$A “gene415” “gene463” “gene179” “gene526” “gene195” “gene938” “gene818″~~のような、ランダムなgene情報が割り当てられたlistを作成
#3 listのxをUpSetRで使用するため、fromList()を使用。nsets =表示サンプル数
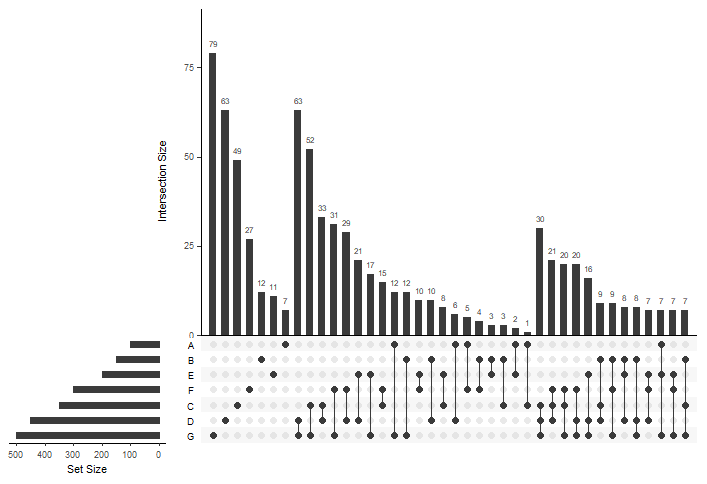
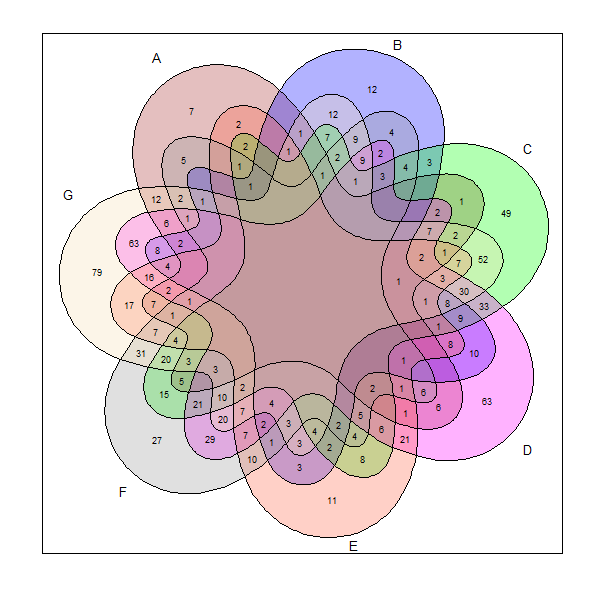
ベン図と比べると、どうでしょうか?

7サンプルの表示をみると、UpSet図の方が視覚的には捉えやすい、でしょうか。。
図の色を変えて、より見やすくしたり、
棒グラフのIntersection sizeを大きい順に並び変えたりしてみます。
upset(fromList(x), nsets = 7, order.by = "freq", point.size = 3, line.size = 1, main.bar.color = "darkblue", sets.bar.color = "lightseagreen") #4 棒グラフの色を指定、因子数の大きい順に並べる
upset(fromList(x[1:7]), nsets = 7, order.by = "freq", point.size = 3, line.size = 1, sets.bar.color = "lightseagreen", query.legend = "bottom",
queries = list(
list(
query = intersects,
params = list("D", "E", "G"),
color = "tomato",
active = T,
query.name = "DEG"))) #5 強調したいところだけ色指定
#4 main.bar.color = で棒グラフの色を指定、sets.bar.color = でSet sizeの棒グラフの色を指定。order.by = 因子数で並べ替え。point.size = , line.size = で下のプロットサイズや線のサイズを指定。
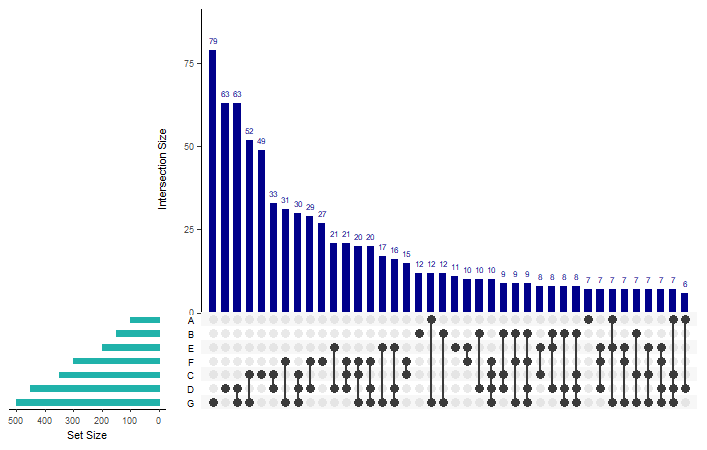
#5 query.legend =で強調色の凡例(queriesのlistの中のquery.name = )場所を指定。queries = list( list(で、強調したい箇所を指定。
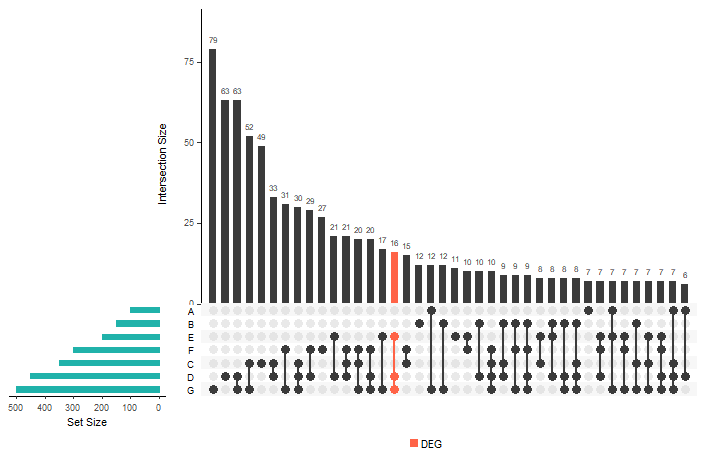
色を用いて、より理解しやすい図を作ることができました。
DとE, Gの3つのサンプルに共通する因子について、色を変えて強調できています。
2017年にUpSetRのコードが報告され、いろいろな分野で使用されてきています。
例えば、Pubmed 検索で、”UpSetR”が2024年2月15日時点で11件ヒットします。
25集合(サンプル)をならべているもの(※2)もあり、多くのサンプルの関係性をみたいときには使いやすいように思いました。
※2 参考文献
Kim KR, Yu JN, Hong JM, Kim SY, Park SY. Genome Assembly and Microsatellite Marker Development Using Illumina and PacBio Sequencing in the Carex pumila (Cyperaceae) from Korea. Genes (Basel). 2023 Nov 10;14(11):2063. doi: 10.3390/genes14112063.
参考webサイト
https://www.r-bloggers.com/2019/04/set-analysis-a-face-off-between-venn-diagrams-and-upset-plots/
https://doi.org/10.1093/bioinformatics/btx364
https://github.com/lgellis/MiscTutorial/tree/master/sets



コメント