タンパク質科学において長らく信じられてきた「構造が機能を決定する」という定説は、21世紀に入り修正を迫られました。ヒトプロテオームの約30〜50%を占めると言われる 天然変性領域 (Intrinsically Disordered Regions; IDRs) の存在です。これらは特定の立体構造を持たず、ふらふらとした紐のように存在していると考えられています。そのため、AlphaFold1, 2などのタンパク質構造予測では、IDRの構造・機能を推定することは困難です。
NARDINI+は、IDRをその一次構造から分類できることを示しています。また、NARDINI+をヒト全タンパク質のIDRsに適用し分類したGINも公開されています。
NARDINI+ Paper: Ruff K.M. et al. (2025) Molecular grammars of predicted intrinsically disordered regions that span the human proteome. Cell. S0092-8674(25)01191-2. doi: 10.1016/j.cell.2025.10.019.
NARDINI+ Github:RUFF_KING_Grammars_of_IDRs_using_NARDINI+, https://github.com/kierstenruff/RUFF_KING_Grammars_of_IDRs_using_NARDINI-
1. 研究(論文とリポジトリ)の背景と目的
近年、Google DeepMind社のAlphaFold1, 2などがタンパク質の立体構造予測で高い性能を発揮しています。ただし、IDRに関しては、ランダムな予測が低いpLDDTスコアで示されるというのが典型的な挙動のようです。これをIDRの有無の予測に使う試みもあるようですが、いずれにせよ、その「性質」や「機能」を推定することはできません。
一方で、IDRは細胞内シグナル伝達や、液-液相分離 (LLPS) による核小体などの非膜オルガネラ形成においてを機能することがわかってきています。
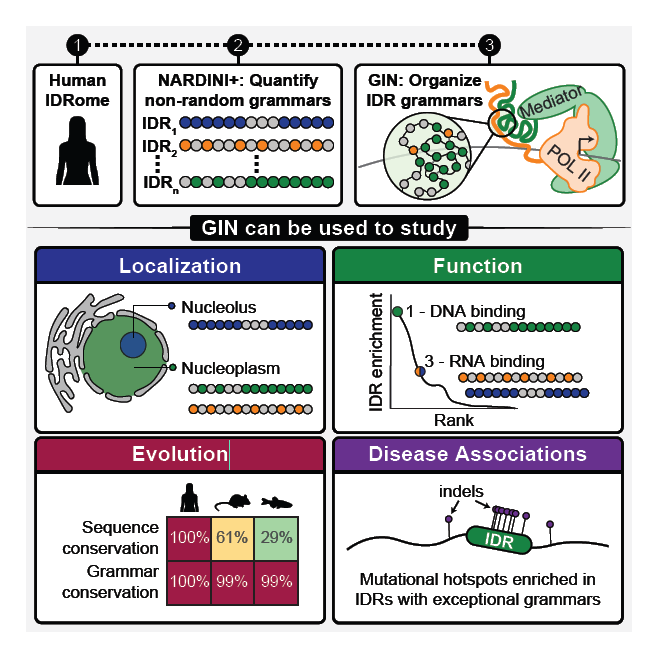
本論文では、IDRのアミノ酸配列はランダムではなく、「どのアミノ酸が多いか(組成)」や「どのように並んでいるか(パターニング)」に意味(文法)があると考え、それを「分子グラマー (Molecular Grammar)」と呼んでいます。
Graphical Abstract
引用 NARDINI+ Paper
今回紹介するRuffらの研究(Washington University in St. Louis)は、この分子グラマーを定量化するアルゴリズム 「NARDINI+」 を開発し、ヒトプロテオーム内の24,508個のIDRを解析しました。その結果、IDRは30種類のクラスター(GINクラスター)に分類でき、それぞれのクラスターが細胞内の「住所(核小体や核スペックルなど)」や「機能」をコードしていることを明らかにしました。
本記事では、GIN (Grammars Inferred using NARDINI+) と、その解析に用いられたPythonコードについて、リポジトリの中身を紐解きながら紹介します。
2. 論文のメソッドとリポジトリ主要ファイルの概要
本研究では、IDRの配列から特徴量を抽出し、教師なし学習によるクラスタリング、分類を試みています。論文の主要なメソッドと、それに対応するGitHubリポジトリのコードは以下のようになっています。
2.1 分子グラマーの定量化:NARDINI+ アルゴリズム
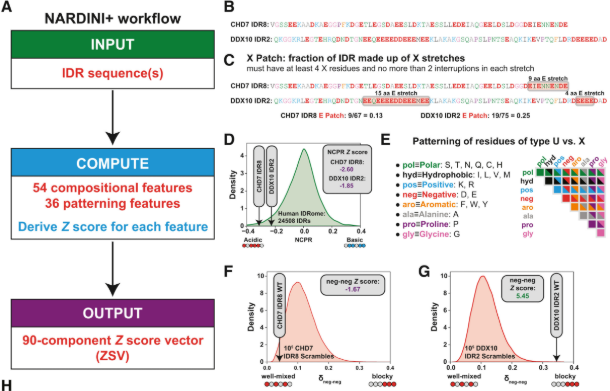
IDRの特徴を数値化するために NARDINI+ というアルゴリズムが提唱されています。これは、アミノ酸組成(54次元)と、配列のパターニング(36次元)を組み合わせた90次元のベクトル(Zスコアベクトル)としてIDRを表現する手法です。
- 組成特徴量: 電荷(FCR, NCPR)、疎水性、特定のアミノ酸の割合など。
- パターニング特徴量: 正電荷と負電荷が混ざっているか、ブロック状に分かれているか、など(例:
pos-neg、pol-hyd)。
これに対応する処理は、リポジトリの 0_Creating_GIN_Clusters ディレクトリに含まれています。
0_extract_GIN_clusters_all_IDRs.ipynb: ヒトの全IDR配列を読み込み、localciderライブラリなどを用いて物理化学的特徴量を計算しています。ここで、各IDRに対して90次元のZスコアベクトル(ZSV)が生成されます。
2.2 ヒトIDRの周期表:GINクラスターの構築
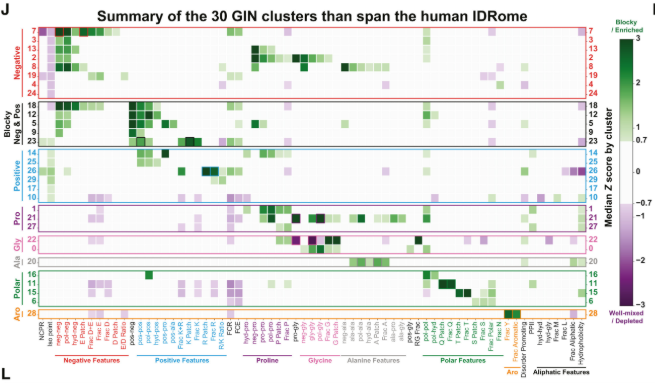
得られたZスコアベクトルを用いて、著者らはK-means法によるクラスタリングを行いました。その結果、ヒトのIDRは30個の離散的なグループ(GINクラスター)に分類できることが分かりました。これは言わば、IDRの「周期表」のようなものです。
このクラスタリングと検証は、以下のコードで実行されています。
0_extract_GIN_clusters_IDRs_100_300.ipynb: 長さ100〜300残基のIDRを抽出してK-meansクラスタリングを行い、最適なクラスター数(K=30)を決定しています。エルボー法やシルエット係数を用いた検証もここで行われています。1_analyze_GIN_clusters_IDRs_100_300.ipynb: 各クラスターがどのような特徴(例:クラスター23は「リジン・ブロック」、クラスター7は「酸性トラクト」)を持っているかを詳細に分析し、ヒートマップとして可視化しています。
2.3 細胞内局在と機能の予測
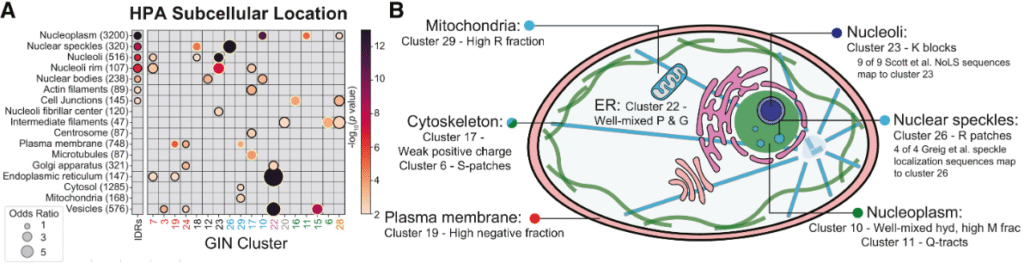
論文の興味深い発見の一つは、「IDRのクラスターが細胞内の住所(局在)を決めている」 という点です。例えば、リジンのブロックを持つ「クラスター23」は核小体に、アルギニンのパッチを持つ「クラスター26」は核スペックルに局在することが示されました。
この解析は、2_Assessing_Enrichment_... および 3_Nucleolus_Speckles_... ディレクトリのコードで行われています。
1_bsp_analyze_IDRs_loc_func_proc_enrichment.ipynb: Human Protein Atlas (HPA) のデータと統合し、フィッシャーの正確確率検定を用いて、各クラスターがどの細胞内局在やGO Term(機能)に濃縮されているかを統計的に解析しています。4_get_HPA_enrichment_by_cluster.ipynb: IDRクラスターと細胞内局在の関連性をバブルプロットなどで可視化するコードです。
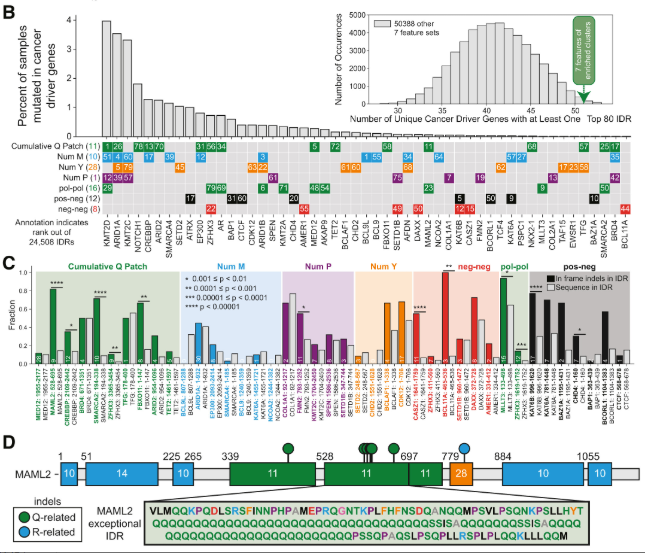
2.4 疾患変異と「例外的なグラマー」
さらに、論文ではがんドライバー遺伝子や融合タンパク質(Fusion Proteins)についても解析しています。がんに関連する変異は、IDRの中でも特に特徴的な(Zスコアが高い)領域、すなわち「例外的なグラマー」を持つ場所に集中していることが示されました。
6_Grammars_in_Cancer/6_bsp_analyze_inframe_indels_COSMIC.ipynb: COSMICデータベースの変異情報を用い、例外的なIDRにおいてインフレーム挿入・欠失が有意に濃縮しているかを検証しています。1_bsp_analyze_fusion_proteins.ipynb: 融合タンパク質において、IDRがどのように「獲得」あるいは「喪失」し、それがグラマーをどう変化させたかを定量化しています。
3. 使用例
Google Colabノートブックが用意されており、自分の興味のあるタンパク質について解析することができます。
例えば、特定のタンパク質(例:FUSやUBTF)のIDRがどのクラスターに属するかを知りたい場合、以下のような手順で解析が可能です(Google Colabでの実行を想定)。
- 遺伝子名または配列の入力: ノートブック上で、解析したいタンパク質の遺伝子名(UniProt ID)リスト、またはFASTA形式の配列を入力します。
- NARDINI+解析の実行: バックグラウンドでPythonスクリプトが走り、組成特徴量とパターニング特徴量が計算され、Zスコア化されます。
- GINクラスターの判定: 計算されたZスコアベクトルと、事前に定義された30個のクラスター重心との距離が計算され、最も近いクラスターが割り当てられます。
出力結果として、以下のような情報が得られます(リポジトリのREADMEサマリより)。
- Cluster Number: そのIDRが属するGINクラスター番号(例:Cluster 23)。
- Z-score Vector: そのIDRの「個性」を表す数値ベクトル。
- Visualizations: IDRの位置や特徴量を可視化したヒートマップや概略図。
これにより、実験をする前に「この変異を入れたらクラスターが変わってしまい、細胞内局在が変化するかもしれない」といった予測を立てることが可能になります。
4. まとめと将来展望
- 配列からグラマーへ: IDRは無秩序ではなく、アミノ酸の並び順(グラマー)に機能情報がコードされている。
- 30のクラスター: ヒトのIDRは30種類のタイプに分類でき、それぞれが特定の細胞内局在(住所)や機能と紐付いている。
- コードの公開: Pythonリポジトリにより、誰でも自分のタンパク質のIDRグラマーを解析可能。
将来展望: この研究は、IDR研究を「定性的な記述」から「定量的な予測・設計」へと押し上げるものです。 今後は、EvoDiff3 のようなタンパク質生成AIと組み合わせることで、「特定の細胞内局在(例:核小体)に運ばれるようなIDRを人工的に設計する」といった合成生物学的な応用や、がんや神経変性疾患で見られるIDRの異常(グラマーの書き換え)を標的とした、新しい創薬アプローチへの展開も期待されます。
IDRの「言語」を理解することで、生命システムの制御に一歩近づけるかもしれません!
参考文献:
1. Jumper, J. et al. (2021). Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 doi: 10.1038/s41586-021-03819-2
2. Abramson, J. et al. (2024) Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature 630, 493–500. doi: 10.1038/s41586-024-07487-w
3. Alamdari, S. et al. (2024) Protein generation with evolutionary diffusion: Sequence is all you need, bioRxiv, 2024–11. doi: 10.1101/2023.09.11.556673


コメント