今回の記事では、タンパク質の全原子生成AIである BoltzGen を実際に実行してみました。BoltzGenのプレプリントとリポジトリでは、タンパク質に対するバインダーや抗体デザインなど、様々な実施例とその設定Yamlファイルが紹介されていますが、ここでは、低分子に対する結合タンパク質の生成を行ってみました。
BoltzGen paper: Stark, H. et al. BoltzGen:Toward Universal Binder Design (2025)
chrome-extension://efaidnbmnnnibpcajpcglclefindmkaj/https://hannes-stark.com/assets/boltzgen.pdf
BoltzGen Github: Boltzgen https://github.com/HannesStark/boltzgen
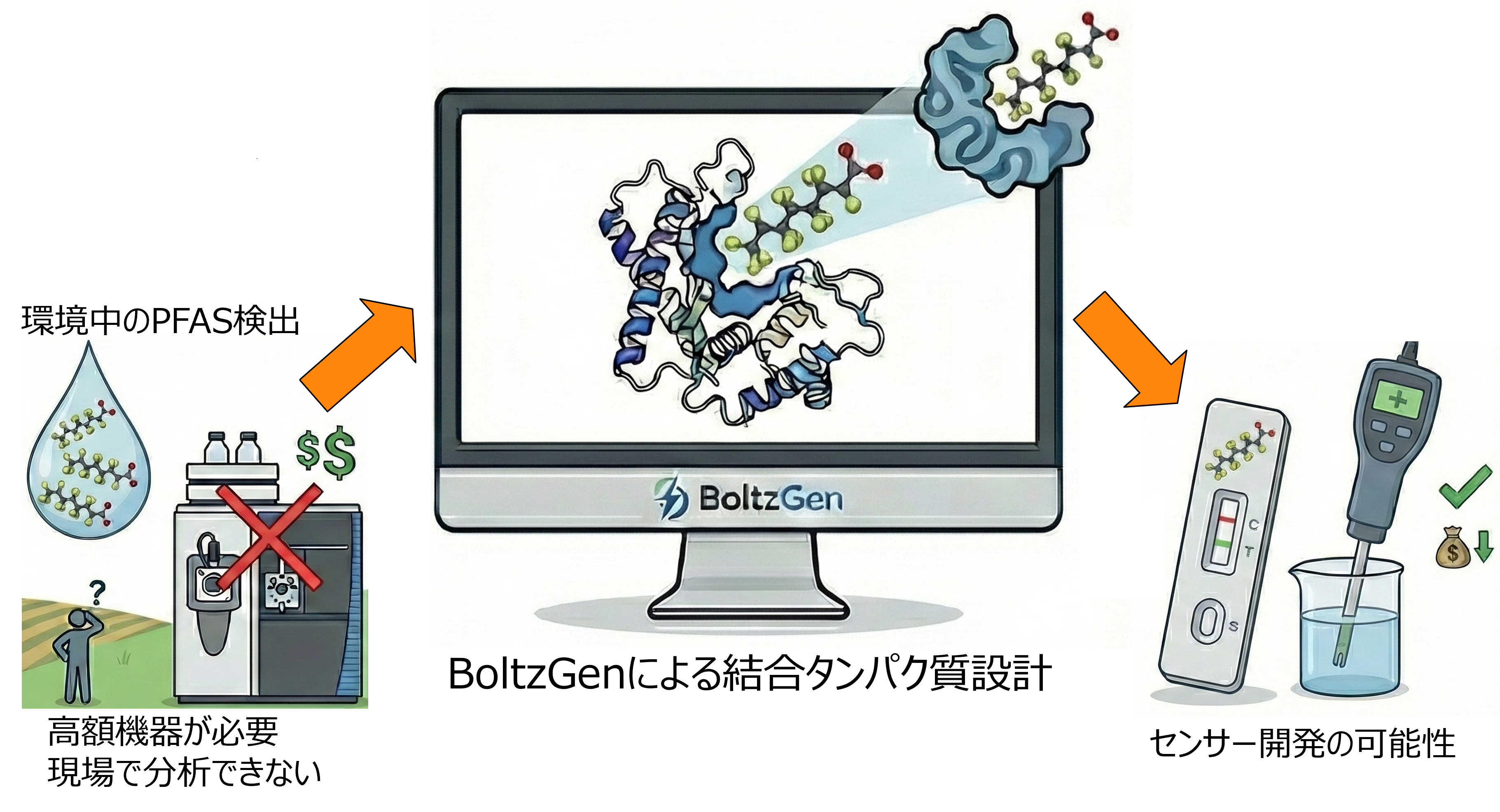
具体的には、タンパク質デザインを環境技術に応用できないか? ということで、ターゲットとするのは、発がん性が疑われ世界中で規制が強化されている有機フッ素化合物、通称PFASです。通常、PFASの検出にはLC-MS/MS等の質量分析が必要です。もし「PFASだけを特異的に捕捉する小さなタンパク質」を設計できれば、安価なセンサーや試薬の開発が可能になるかもしれません。
本記事では、BoltzGenの「低分子結合(Small Molecule Binding)」を使って、この課題に挑戦してみました。チュートリアル的な何かになれば幸いですm(_ _”m)
1. ターゲットの準備
PFASの一種であるPFOA(ペルフルオロオクタン酸)をターゲットにしました。BoltzGenでは、タンパク質だけでなく、低分子もターゲットに設定することが可能で、Chemical Component Dictionary (CCD) codes か SMILES で、低分子構造を指定します。
ここでは、SMILES記法でPFOA構造を準備しました。以下のPubchem、Perfluorooctanoateの情報を利用しました。https://pubchem.ncbi.nlm.nih.gov/compound/Perfluorooctanoate
PFOA SMILES: C(=O)(C(C(C(C(C(C(C(F)(F)F)(F)F)(F)F)(F)F)(F)F)(F)F)(F)F)[O-]
今回は、この低分子を「リガンド」として定義し、それを包み込むポケットを持つタンパク質を設計するということです。
2. 設定ファイル(YAML)の作成
PFOAをリガンドとして結合するタンパク質を生成させる設定で、YAMLファイルを作りました。PFOAの構造と、その他は何残基~何残基までのタンパク質を生成するか、その範囲だけ設定し、保存する配列や結合残基などは指定しません。
以下の内容を design_pfas_sensor.yaml として、boltzgen/example に保存しました。
# design_pfas_sensor.yaml
entities:
# 1. ターゲット低分子(PFOA)の定義
- ligand:
id: B # リガンドのID
smiles: "C(=O)(C(C(C(C(C(C(C(F)(F)F)(F)F)(F)F)(F)F)(F)F)(F)F)(F)F)[O-]" # PFOA
# 2. バインダー(センサー素子)の設計
- protein:
id: A
sequence: 80..180 # 残基数
設定のポイント:
– ligand: smiles: もしくはccd: を指定することで、リガンドとして設定されます。
– protein:
sequence: 80..180 生成させるアミノ酸長の範囲を指定しています。小型のタンパク質を設計することで、大腸菌等での大量発現を容易にしたいという狙いです。タンパク質の生産量が多ければ、応用の面で有利です。
3. 計算の実行
boltzgen ディレクトリで以下のコマンドを実行すると、workbench/test_pfas ディレクトリが作成されて、計算結果が保存されていきます。
boltzgen run example/pfas/design_pfas_sensor.yaml \
--output workbench/test_pfas \
--protocol protein-small_molecule \
--num_designs 1000 \
--budget 10*リポジトリ記載の情報によれば、良いデザインタンパク質を得るためには、10,000〜60,000配列くらいを生成させる必要があるとのことです。–num_designs で指定できます。今回はお試しということで、1000個の配列を生成させました。また、–budget 10として、スコアの上位10個を最終的なデザインとしました。
以下は、計算開始時に作成されるsteps.yamlファイルの内容ですが、低分子への結合タンパク質生成のタスクの際に実施される、Boltz-2を用いた結合親和性予測(affinity.yaml)が入り、7つのステップで最終的な結果が得られます。
steps:
- name: design
config_file: config/design.yaml
- name: inverse_folding
config_file: config/inverse_folding.yaml
- name: folding
config_file: config/folding.yaml
- name: design_folding
config_file: config/design_folding.yaml
- name: affinity
config_file: config/affinity.yaml
- name: analysis
config_file: config/analysis.yaml
- name: filtering
config_file: config/filtering.yamlGeForce RTX 2080 Ti を積んだパソコンで、1日弱くらいずっと計算が続きましたが、完走できました!
4. 結果
計算が終了したら、all_designs_metrics.csv、final_designs_metrics_10.csv、results_overview.pdf などのファイルが作成されます。これらに、全デザイン(1000個)とランク10位までのアミノ酸配列や様々な予測値、それらの統計値が記載されています。
今回の全1000個のデザインと、選定されたトップ10デザインの集計統計を比較すると、以下のようになっており、フィルターが機能することで、良さそうなデザインが選抜できているようです。
| メトリクス名 | 全デザイン平均 (N=1000) | トップ10デザイン平均 (N=10) |
num_design (残基数) | 151.6 | 127.8 |
filter_rmsd | 2.822 | 1.021 |
designfolding-filter_rmsd | 3.097 | 1.246 |
affinity_probability_binary1 | 0.180 | 0.617 |
design_to_target_iptm | 0.752 | 0.845 |
min_design_to_target_pae | 3.111 | 1.828 |
plip_hbonds_refolded | 0.744 | 1.600 |
delta_sasa_refolded | 418 | 437 |
トップ10デザインは、全デザインと比較して、filter_rmsd, designfolding-filter_rmsdが低く、構造精度に優れており、affinity_probability_binary1 や design_to_target_iptm の値が高く、結合する確率が高そうです。ターゲットとの相互作用に関する指標も高そうです。
最終的な立体構造ファイル(.cif)が、final_10_designs ディレクトリに保存されています。ランクトップrank0001の構造をPyMOLで読み込むと、以下のようにPFOAを内部に収めたタンパク質の構造が確認できました!
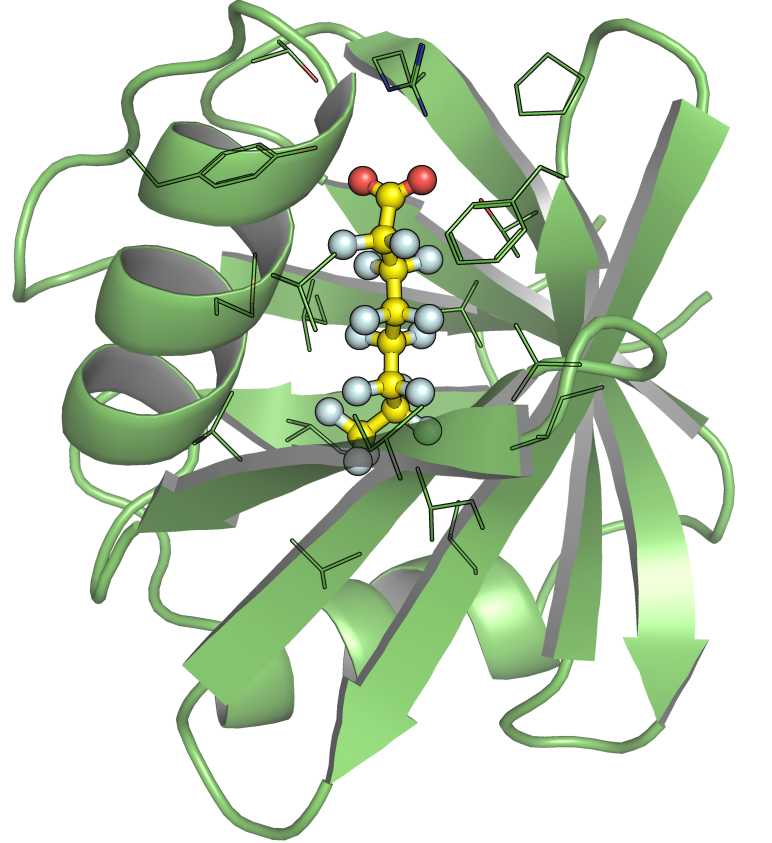
βシートがバレルようになりつつ、αヘリックスと合わせ技でPFOAを包んでいるような形になっています。有機フッ素と直接接触する部位には疎水性アミノ酸側鎖が配置されています。また、PFOAのカルボキシル基は分子表面に露出するような形となり、それをアルギニン側鎖が抑えているようにみえます。
今回は、まだ1000個を設計した段階ですが、良さそうな結果です!
PyMOLは過去に記事でも取り上げておりますので、参照ください。
5. まとめと今後の展望
「低分子バインダー設計」の手法は、医薬をはじめ、様々な分野で期待されています。今回の記事では、BoltzGenを用いて、環境汚染物質を結合するタンパク質の設計を試みました。
BoltzGenはインストールや計算設定も使いやすく、設計とフィルタリングも連続で実行され、大変素晴らしいです。抗体デザインやタンパク質へのバインダー設計と併せて、これまで物性やコスト等の問題でターゲットにできなかった物質に対するバインダー設計が実現可能となっているかも。AIによるタンパク質設計、さらなる発展に期待ですね!




コメント