以前の投稿で、熊本県の新型コロナ陽性者数動向と他都道府県との相関関係について書きました。
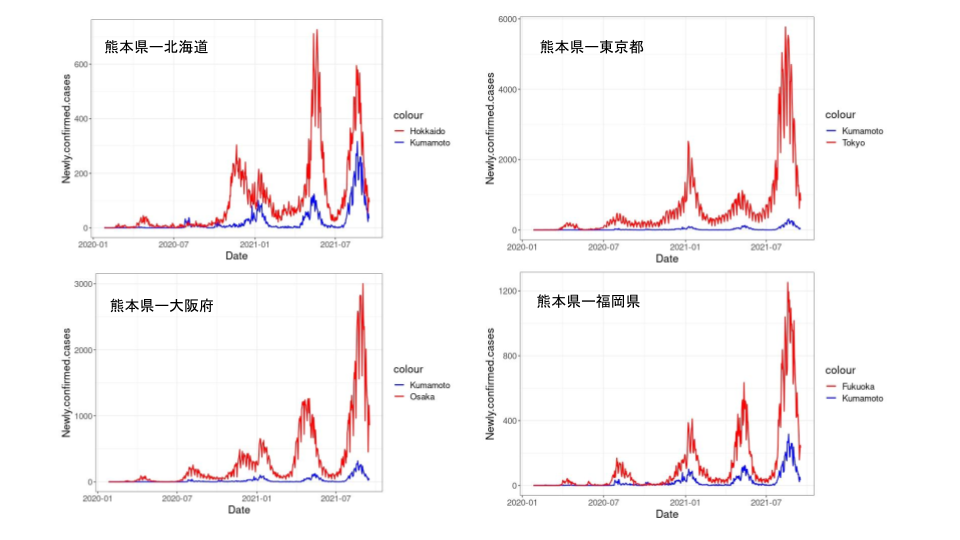
厚労省のオープンデータを用いて、引き続きデータ解析を行います。
#1 #2まで以前の投稿と同じ部分。
patient_prefecture<-read.csv("newly_confirmed_cases_daily.csv") #厚労省のwebから入手したcsvをそのまま読み込む
patient_Hokkaido<-subset(patient_prefecture, Prefecture=="Hokkaido") #北海道のデータ
patient_Aomori<-subset(patient_prefecture, Prefecture=="Aomori") #青森県のデータ
patient_Iwate<-subset(patient_prefecture, Prefecture=="Iwate") #岩手県のデータ
patient_Miyagi<-subset(patient_prefecture, Prefecture=="Miyagi") #宮城県のデータ
・・・全都道府県データ作成
patient_Hokkaido2<-patient_Hokkaido[, c(-2)] #北海道のデータ
Hokkaido<-patient_Hokkaido2$Newly.confirmed.cases #北海道の人数だけのデータ
patient_Aomori2<-patient_Aomori[, c(-2)]
Aomori<-patient_Aomori2$Newly.confirmed.cases
patient_Iwate2<-patient_Iwate[, c(-2)]
Iwate<-patient_Iwate2$Newly.confirmed.cases
patient_Miyagi2<-patient_Miyagi[, c(-2)]
Miyagi<-patient_Miyagi2$Newly.confirmed.cases
・・・全都道府県データ作成
#2 ここまで
#3
A_list<-cbind(Hokkaido,Aomori,Iwate,Miyagi,Akita,Yamagata,Fukushima,Ibaraki,Tochigi,
Gunma,Saitama,Chiba,Tokyo,Kanagawa,Niigata,Toyama,Ishikawa,Fukui,Yamanashi,Nagano,
Gifu,Shizuoka,Aichi,Mie,Shiga,Kyoto,Osaka,Hyogo,Nara,Wakayama,Tottori,Shimane,Okayama,
Hiroshima,Yamaguchi,Tokushima,Kagawa,Ehime,Kochi,Fukuoka,Saga,Nagasaki,Kumamoto,Oita,
Miyazaki,Kagoshima,Okinawa) #北海道〜沖縄県のデータを一つにまとめた
Okinawa Kagoshima Miyazaki Oita Kumamoto Nagasaki Saga Fukuoka Kochi Ehime Kagawa
[1,] 0 0 0 0 0 0 0 0 0 0 0
[2,] 0 0 0 0 0 0 0 0 0 0 0
[3,] 0 0 0 0 0 0 0 0 0 0 0
[4,] 0 0 0 0 0 0 0 0 0 0 0
[5,] 0 0 0 0 0 0 0 0 0 0 0
[6,] 0 0 0 0 0 0 0 0 0 0 0
Tokushima Yamaguchi Hiroshima Okayama Shimane Tottori Wakayama Nara Hyogo Osaka
[1,] 0 0 0 0 0 0 0 0 0 0
[2,] 0 0 0 0 0 0 0 0 0 0
[3,] 0 0 0 0 0 0 0 1 0 0
[4,] 0 0 0 0 0 0 0 0 0 1
[5,] 0 0 0 0 0 0 0 0 0 0
[6,] 0 0 0 0 0 0 0 0 0 0
Kyoto Shiga Mie Aichi Shizuoka Gifu Nagano Yamanashi Fukui Ishikawa Toyama Niigata
[1,] 0 0 0 1 0 0 0 0 0 0 0 0
[2,] 0 0 0 0 0 0 0 0 0 0 0 0
[3,] 0 0 0 1 0 0 0 0 0 0 0 0
[4,] 0 0 0 0 0 0 0 0 0 0 0 0
[5,] 1 0 1 0 0 0 0 0 0 0 0 0
[6,] 0 0 0 0 0 0 0 0 0 0 0 0
Kanagawa Tokyo Chiba Saitama Gunma Tochigi Ibaraki Fukushima Yamagata Akita Miyagi
[1,] 0 0 0 0 0 0 0 0 0 0 0
[2,] 0 0 0 0 0 0 0 0 0 0 0
[3,] 0 0 0 0 0 0 0 0 0 0 0
[4,] 0 0 0 0 0 0 0 0 0 0 0
[5,] 0 0 1 0 0 0 0 0 0 0 0
[6,] 0 0 0 0 0 0 0 0 0 0 0
Iwate Aomori Hokkaido
[1,] 0 0 0
[2,] 0 0 0
[3,] 0 0 1
[4,] 0 0 0
[5,] 0 0 0
[6,] 0 0 0
#4
library(corrplot) #ここではsuperheatを採用。
corrplot(cor(A_list), col= colorRampPalette(c('white', 'white', '#007FFF', '#00007F'))(100), is.corr = FALSE,tl.col="black",tl.cex=0.8)
#相関係数をヒートマップに。
#1-#2は以前の投稿にも記載しています。各都道府県の1日当たりの陽性者数情報のみのデータにしました。
#3 cbind(A, B, C)AとBとCの列を横に結合させます。北海道〜沖縄県のデータを並べました。
#4 cor(A_list) A_listの各都道府県の数値変動同士の相関係数を求めます。
これをcorrplotでヒートマップにして表しました。
詳細について以下の投稿で相関行列について説明しています。
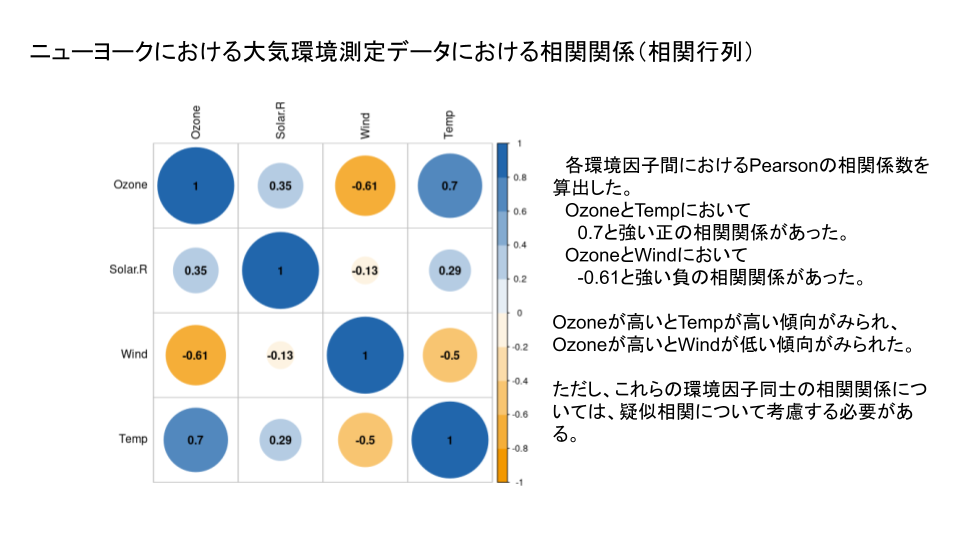
col= colorRampPalette(c(相関係数の一番小さい数値〜大きい数値))(色指定の分割数) は色を指定しています。
変化が分かりやすい形で色を設定しました。
is.corr = FALSE, デフォルトは is.corr = TRUEであり、相関係数-1〜1で色やlegend表示されます。
これをFALSEにすることで、このデータの相関係数の最も低い値〜最も高い値で、色とlegendを表示されます。
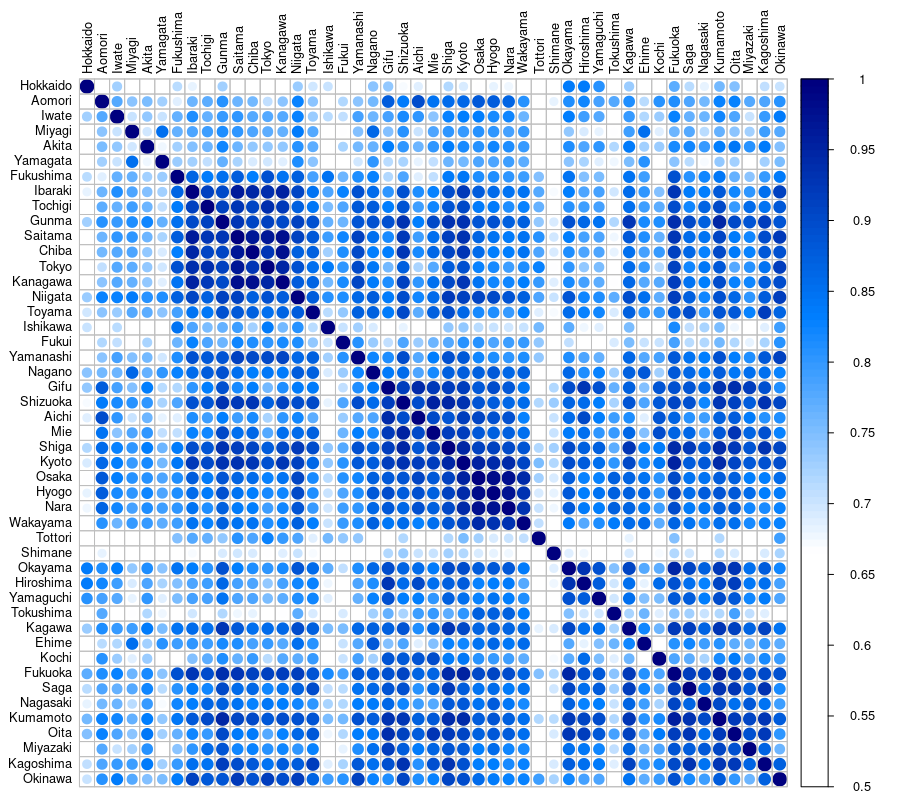
都道府県が北から南へと並べており、相関係数の大小は青さで示しています。
例えば、最も上に配置している北海道の行を横に見ると、各都道府県との相関係数の大小がわかります。北海道はどの都道府県とも色が薄く、相関が低いことがわかります。やや岡山県、広島県、山口県等との相関が高いようです。
また、関東地方、中部地方、関西地方、九州地方あたりは、その地域ごとにかたまりとなって色が濃く、相関が高い傾向が見られました。近隣の都道府県と相関が高い、つまり新規陽性者発生動向が似ていることがみてとれました。
一方、東北地方や、鳥取県や島根県においては、周辺の県との相関も低い傾向が確認されました。
東京、大阪、福岡等、大都市圏からその周辺県へ影響が及んでいることを示しているのかもしれません。相関関係と因果関係は分て考えないといけませんし、疑似相関をみている可能性もありますが、大変興味深い結果でした。


コメント