ベン図は、集合論や統計学で広く使用され、重なりや包含関係を視覚的に理解するのに役立ちます。
異なるグループの要素の交わりや固有の特性を把握し、データの関連性を把握するのにとても便利です。
1. ベン図とは
2. ベン図用ダミーデータ作成(遺伝子情報)
3. ベン図 遺伝子数による色の濃淡指定 ―ggVennDiagram(最大7サンプル表示)―
4. ベン図 サンプル別色指定 ―ggvenn(最大4サンプル表示), venn(最大7サンプル表示)―
ベン図は、複数の集合やグループの要素の重複や共通性を視覚的に表現するための図です。
円や楕円などの形状を用い、それぞれの形状が異なる集合やグループを表します。
各形状の重なり合いや共通部分が、対応する集合やグループの要素の共通性を示します。
通常、2つ以上の集合が組み合わさったり交わったりする場合に使用され、データの重複や相互関係を明示的に表示します。

遺伝子情報のダミーデータを作成しましょう。
genes <- paste("gene",1:1000,sep="") #1
set.seed(0123)
x <- list(A=sample(genes,100),
B=sample(genes,150),
C=sample(genes,350),
D=sample(genes,450),
E=sample(genes,200),
F=sample(genes,300),
G=sample(genes,500)) #2 サンプル数は7設定
x #3
#$A
# [1] "gene415" "gene463" "gene179" "gene526" "gene195" "gene938" "gene818" "gene118" "gene299"
# [10] "gene229" "gene244" "gene14" "gene374" "gene665" "gene602" "gene603" "gene768" "gene709"
# [19] "gene91" "gene953" "gene348" "gene649" "gene355" "gene840" "gene26" "gene519" "gene426"
# [28] "gene979" "gene766" ~~
#$B
# [1] "gene286" "gene908" "gene671" "gene121" "gene110" "gene158" "gene64" "gene483"
# [9] "gene910" "gene477" "gene480" "gene711" "gene67" "gene663" "gene890" "gene847"
# [17] "gene85" "gene165" "gene648" "gene51" "gene74" "gene178" "gene362" "gene236"
# [25] "gene610" "gene330" ~~
#1 paste関数は文字列を作成。”gene”と1から1000までの整数を結合して、”gene1″, “gene2”, …, “gene1000” のような文字列ベクトルを生成。
#2 list関数は、指定された要素を持つリストを作成。
例えば、A=sample(genes,100)は、遺伝子名のベクトル “genes” からランダムに100個の要素を選んで、集合Aに割り当て。B以降も同様。
#3 x内で、A~Gにランダムにgeneが割り当てられていることを確認。
それでは、この遺伝子情報のダミーデータを用いてベン図を作成していきます。
install.packages("ggVennDiagram")
install.packages("ggplot2")
library(ggVennDiagram)
library(ggplot2)
#各領域に属するgene数を表示
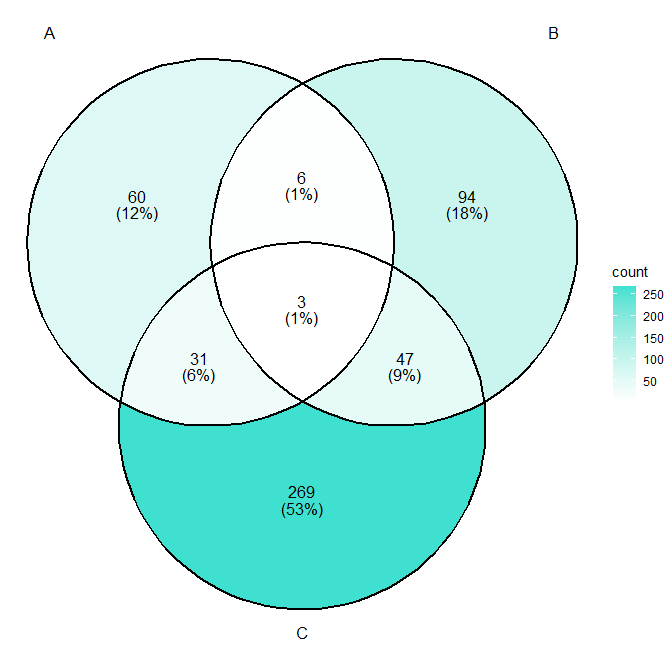
ggVennDiagram(x[1:3]) #4 A~Cまでのデータを用いてベン図を作成
ggVennDiagram(x[1:3],label_alpha=0) + scale_fill_gradient(low="white",high = "turquoise") #5
#4 ベン図の作成 それぞれの領域に含まれるgeneの数(割合)に応じた色の表示。

#5 ベン図の数表示の背景を削除(label_alpha=0)し、色表示を指定(+ scale_fill_gradient)。
色は、Rカラーコード 参考にしていただけたらと思います。
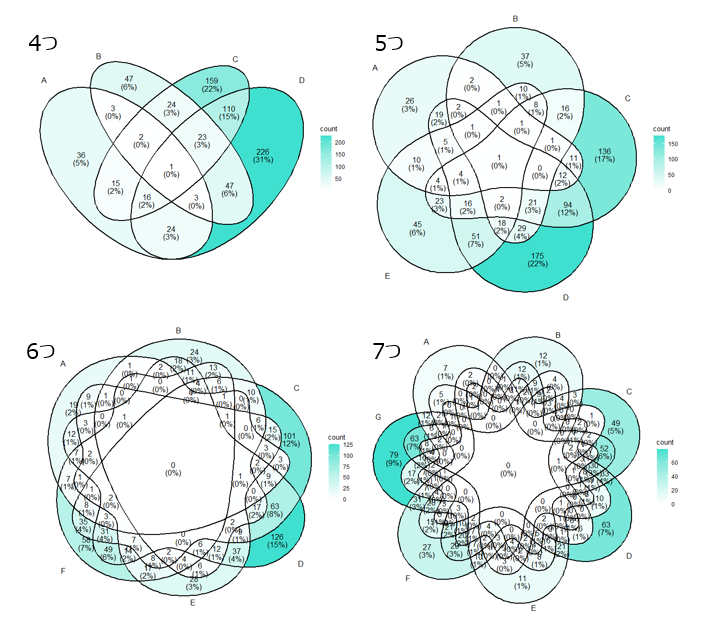
ベン図は、視認性の観点から3つのサンプルまでの表示が望ましいとされていますが、より多くのサンプル表示で説明されたい場合もあると思います。
ggVennDiagramパッケージでは最大7サンプルまで、ベン図での表示が可能になりました。
これは、最近公開されたvennパッケージからインポートされるようになったためです。
なお、視認性については、使用される目的などに応じて御判断いただけたらと思います。
ggVennDiagram(x[1:4],label_alpha=0) + scale_fill_gradient(low="white",high = "turquoise") #6 4つ ggVennDiagram(x[1:5],label_alpha=0) + scale_fill_gradient(low="white",high = "turquoise") #7 5つ ggVennDiagram(x[1:6],label_alpha=0) + scale_fill_gradient(low="white",high = "turquoise") #8 6つ ggVennDiagram(x,label_alpha=0) + scale_fill_gradient(low="white",high = "turquoise") #9 7つ
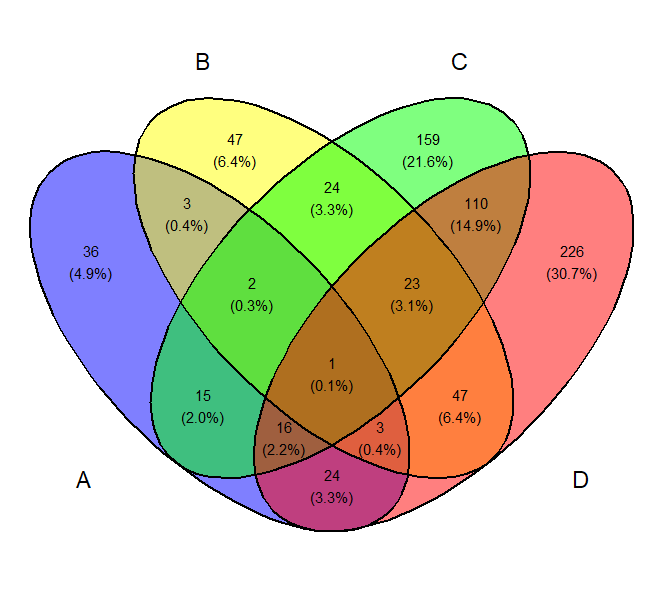
サンプルごとに色を変えた表示方法を見ていきましょう。
install.packages("ggvenn")
library(ggvenn)
ggvenn(x) #10 4サンプルまで表示可
install.packages("venn")
install.packages("ggpolypath")
library(venn)
library(ggpolypath)
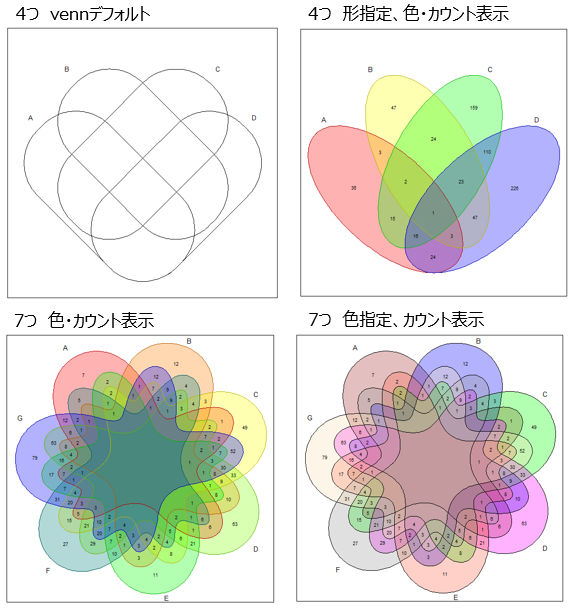
venn(x[1:4]) #11 4つ vennデフォルト
venn(x[1:4], ilabels = "counts", ggplot = TRUE, zcolor ="style",ellipse = TRUE) #12 4つ 形指定、色・カウント表示
venn(x, ilabels = "counts", ggplot = TRUE, zcolor ="style") #13 7つ 色・カウント表示
venn(x, ilabels = "counts", ggplot = TRUE, zcolor =c("brown", "blue", "green", "magenta", "tomato", "grey60", "wheat")) #14 7つ 色指定、カウント表示
#10 ggvenパッケージは、4サンプルまで表示可。このため、5サンプル目以降(E~G)は表示不可。各サンプルごとに異なる色での表示が可能
#11~14は、それぞれ以下の表示。ilabels = “counts”で、各領域の遺伝子情報数を表示。zcolor =で色を指定。styleは既に登録されている色情報を使用。ellipse = TRUEで、楕円形で表示できる。オリジナルの色にしたい場合は、#14のように色を指定。
色は、上記同様Rカラーコード 参考にしていただけたらと思います。
Rでベン図を作成する際の参考になれば幸いです。
ベン図とは違うアプローチのアップセット図(UpSet図)についての記事もかいていますので、ぜひ参考にされてください。

参考
https://cran.r-project.org/web/packages/ggVennDiagram/ggVennDiagram.pdf
https://cran.r-project.org/web/packages/venn/venn.pdf
https://venn.bio-spring.top/using-ggvenndiagram
https://github.com/gaospecial/ggVennDiagram




コメント