タンパク質は生命活動を担う多機能な生体分子であり、その機能の多くはタンパク質間相互作用(Protein-protein interactions:PPIs)によって発揮されています。このPPIsを標的として阻害や制御できるタンパク質バインダーの設計は、薬剤開発、診断、そしてバイオテクノロジー分野において新たな可能性を切り開くものと期待されています。
※この記事の作成には、文章の校正の一部で生成AIを活用しています。最終的な内容は、全て筆者が確認・編集しています。
1. 論文の背景とリポジトリの関連性
論文:Pacesa M., Nickel, L., Schmidt, J., Pyatova, E., Schellhaas, C., Kissling, L., et al., (2024). BindCraft: one-shot design of functional protein binders, bioRxiv 2024.09.30.615802; doi: 10.1101/2024.09.30.615802
※アクセプト論文(2025/8/27公開) Pacesa, M., Nickel, L., Schellhaas, C. et al. One-shot design of functional protein binders with BindCraft. Nature (2025). doi:10.1038/s41586-025-09429-6
GitHub(リポジトリ):BindCraft https://github.com/martinpacesa/BindCraft
従来のタンパク質バインダーの計算設計は、ヒット効率が低く、実際に機能するバインダーを得るためには、大規模なin vitroスクリーニングを必要とします。また、天然の結合部位が知られていないターゲットに対する設計は、特に難しいと考えられています。
BindCraftは、Google DeepMindが開発したタンパク質構造予測モデルAlphaFold2 (AF2)の能力を最大限に活用することで、in vitroスクリーニングや実験的最適化なしに、ナノモルオーダーの親和性を有するバインダーを効率的に生成することを目指しています。
GitHubリポジトリでは、BindCraftパイプラインのオープンソース実装が提供されています。このパイプラインを実行することで、コンピュータ上での設計をほぼ自動化でき、実験的検証までの一連のプロセスを大幅に加速することが期待されます。
2. リポジトリの概要・背景
BindCraftリポジトリは、ColabDesign(※)を中核とし、ProteinMPNN、PyRosettaといった最先端の計算生物学ツールを統合したタンパク質バインダー設計パイプラインの実装です。ユーザーがターゲットタンパク質を指定するだけで、実験的な評価へとつながる十分な数のバインダーデザインを、効率的かつ自動的に生成することを実現しています。
※ColabDesign
ColabDesignは、AF2の訓練済み重みを直接活用するタンパク質設計のフレームワークです。BindCraftの責任著者の一人であるSergey Ovchinnikov氏によって実装が公開されています。
3. 論文Methodsとリポジトリ主要ファイルの概要
論文の主要な設計技術
BindCraftの設計プロトコルは、以下の主要な技術的要素から構成されます。
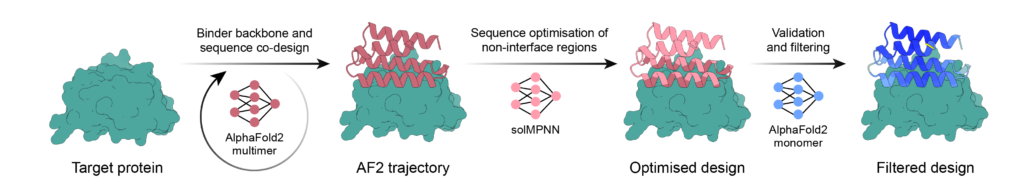
図1: BindCraftバインダー設計パイプライン概要図
出典: GitHub – BindCraft (Copyright 2024 Martin Pacesa)
- 初期設計:AF2 multimerによるバインダー構造と配列の幻覚生成
ターゲットタンパク質に対し、タンパク質複合体の予測に特化して訓練されたAF2 multimerモデルを用いて、バインダーの配列と構造と相互作用面を同時に、以下のような流れで幻覚生成(hallucination)します。まずランダムに生成したバインダーの配列ロジットからAF2が複合体構造を予測し、その予測結果に基づく設計損失(design loss(バインダーのpLDDTや界面のi_pTMなどのAF2の信頼度スコアも含む))を利用して、AF2ネットワークを介して逆伝播によって誤差勾配が生成されます。生成された誤差勾配は、確率的勾配降下法を用いてバインダー配列を最適化し、設計損失を最小化するために、複数回のイテレーションを経て更新されます。これらは、BindCraftが設定する条件下で、ColabDesignライブラリが機能することで、次の4段階で実行されます。
① 連続シーケンス空間での最適化:ロジット入力を利用し、各バインダー位置で複数のアミノ酸を連続的な空間で最適化を行います。合計75イテレーション実施。
② シーケンス確率への正規化:softmax関数を用いて配列ロジットを配列確率に正規化し、設計空間をより現実的な配列表現へと収束させます。45イテレーション実施。
③ ストレートスルー推定器の使用:AF2 multimerモデルでワンホット(離散的)表現を確認しつつ、softmax(連続的)表現を介して逆伝播を実行します。5イテレーション実施。
④ ワンホット離散エンコーディングへの変換と最適化:配列をワンホットの離散エンコーディングに変換することで、離散的な、実際のアミノ酸配列へと収束させます。15イテレーション実施。
単一モデルへの過学習を避けるため、各イテレーションで5つの訓練済みAF2 multimerモデルをランダムに切り替えながら実施されます。 - 配列最適化:MPNNsolによるバインダーコアと表面の最適化
AF2 multimerで幻覚生成されたバインダーは、実験的に低い発現レベルを示すことを考慮し、デザインされたバインダーとターゲットタンパク質との間の結合界面を維持しつつ、バインダーのコアと表面の配列をProteinMPNNの可溶性重み(タンパク質の溶解性を高めるよう訓練されたパラメータセット:MPNNsol)を用いて最適化します。結合親和性に影響を与えることなく、バインダーの安定性や溶解性を向上させることが目的です。 - 再予測と堅牢なフィルタリング:AF2 monomerモデルとRosettaによる評価
最適化されたバインダーは、AF2 monomerモデルで再予測されます。このモデルは単量体タンパク質に特化して訓練されており、タンパク質-タンパク質相互作用の予測バイアスを最小限に抑え、高品質な結合界面を得ることが期待されます。最終的に、AF2の信頼度スコア(pLDDT、pTM、i_pTMなど)と、PyRosettaライブラリを用いた物理化学的な指標(界面形状相補性、界面エネルギー、不飽和水素結合数など)に基づいて、設計がフィルタリングされ、実験テストに適したデザインが選抜されます。
リポジトリ主要ファイルの役割
これらの設計プロトコルは、リポジトリ内の複数のPythonファイルが連携して実現されています。
■bindcraft.py: パイプラインの統括
BindCraftの設計パイプライン全体、前述の「1.初期設計」、「2.配列最適化」、および「3.再予測と堅牢なフィルタリング」という主要な3つの設計フェーズを順序立てて実行し、全体のワークフローを管理します。設定ファイルの読み込み、AF2モデルのロード、結果の保存、進捗監視、フィルタリングと出力の決定など、パイプライン全体の流れを統括します。
■functions/colabdesign_utils.py: AF2/MPNNによる設計と予測の中核
既存のColabDesignライブラリを活用するパイプラインで、AF2およびProteinMPNNの機能を実装し、新規タンパク質バインダーの設計と評価を行います。
ー binder_hallucination関数: 1.初期設計に直接対応する部分で、AF2ベースのモデルを用いてバインダーの配列と構造を幻覚生成します。多様な設計損失(pLDDT、pAE、i_pTM、ヘリシティなど)を利用した逆伝播によって、1.初期設計①~④の4段階の配列最適化を実行します。この関数内に、ColabDesignライブラリを活用した、初期設計①~④に該当する配列最適化の関数等が組み込まれています。
・design_logits関数:① 配列ロジットの連続シーケンス空間での最適化
・design_soft関数:② Softmaxによるシーケンス確率への正規化
・design_hard関数:③ ストレートスルー推定器
・design_pssm_semigreedy関数:④ ワンホット離散エンコーディング
ー mpnn_gen_sequence関数: 2.配列最適化に対応する部分で、具体的には、設計されたバインダー構造を基にProteinMPNNモデルの可溶性の重み(MPNNsol)を用いて複数の候補アミノ酸配列を生成し、バインダーの安定性や溶解性を向上させます。
ー predict_binder_complex / predict_binder_alone関数: 3.再予測と堅牢なフィルタリングに対応する部分です。生成された各バインダー配列の品質を検証するため、AF2ベースのモデルで複合体およびバインダー単独の構造を予測し、pLDDT、pTM、i_pTMなどの詳細な信頼度スコアで評価します。
■functions/pyrosetta_utils.py: 構造解析と最適化
PyRosettaライブラリを利用し、タンパク質構造の物理ベースの詳細な解析とエネルギーの最適化を行うための多機能なユーティリティ群を提供します。3.再予測と堅牢なフィルタリングに対応する部分で、物理化学的なフィルター基準(界面形状相補性、界面エネルギー、不飽和水素結合数など)の計算を行います。具体的には、score_interface関数で界面特性の評価、pr_relax関数で構造のエネルギー的緩和などが実行されます。
■functions/biopython_utils.py: 構造品質と配列の検証
BioPythonライブラリとNumPy、SciPyといった科学計算ツールを駆使し、タンパク質構造設計の品質評価と詳細な解析を行うための多機能なユーティリティ群を提供します。3.再予測と堅牢なフィルタリングに対応する部分で、物理的妥当性と信頼性が多面的に検証されます。
validate_design_sequence関数で配列の健全性、calculate_clash_score関数で構造内のクラッシュ数、calc_ss_percentage関数で二次構造(ヘリックス、シート、ループ)の割合とpLDDTスコアの評価など、設計された構造の物理的妥当性が多角的に検証されます。
以上のように、BindCraftのパイプラインは、bindcraft.pyが全体の流れを制御し、colabdesign_utils.pyがColabDesignライブラリを利用しつつAF2とProteinMPNNを用いた設計と予測を行います。pyrosetta_utils.pyとbiopython_utils.pyが、それぞれPyRosettaとBioPythonの強力な機能を用いて、設計された構造の物理的妥当性と品質を評価します。
4. モデルの検証
BindCraftは、その設計能力を多様な生物学的ターゲットに対して実証し、その実用性の高さを示しました。
・既知の結合部位を持つ細胞表面受容体へのバインダー設計: 免疫シグナル伝達分子であるPD-L1およびIFNAR2に対し、それぞれ615 nMおよび260 nMの平衡解離定数(Kd)を持つナノモル親和性のバインダーを設計することができました。また、それぞれすでに標的部位に結合することが分かっているバインダーと表面プラズモン共鳴(SPR)を使用した競合アッセイを行ったところ、競合が確認されました。
・既知の結合サイトがない部位へのバインダー設計: 構造的に複雑な細胞外ドメイン(ECD)を持つCD45に対し、14.7 nMのKdを持つバインダーを設計することができています。また、円二色性(CD)において予想されたαヘリックスシグナルが観察され、設計の正しいフォールディングが検証されました。
・膜タンパク質へのバインダー設計: タイトジャンクションの完全性維持に重要な膜タンパク質であるクラウディン1を標的としました。膜上のタンパク質を扱うのは難しいため、可溶性アナログ(膜タンパク質の本来の結合部位を保持するように計算設計された可溶性バージョンのクラウディン1)を用いてバインダーが設計されました。食中毒の原因菌であるClostridium perfringensが産生するenterotoxin (CpE) は、クラウディン1の細胞外ドメインに結合し、細胞毒性を示します。設計されたバインダーは、このCpEによる細胞毒性を効果的に阻害することができました。
・アレルゲンへのバインダー設計:アレルゲンは多様なタンパク質で構成され、表面が高度に帯電しているため、一般的に疎水性の結合部位が計算によるバインダー設計に適しているとされる中、アレルゲンはより難しい標的とされていました。しかし、BindCraftは、シラカバ花粉アレルゲンBet v1に対するバインダーの設計に成功しています。設計されたバインダーの1つが、120nMのKdを示し、さらに、その結合相互作用の検証も行われました。さらに、この設計バインダーがシラカバアレルギー患者由来の血清サンプルにおいてIgE結合を効果的に阻害(IgEブロッキングELISAで、最大50%阻害)したことも報告されました。計算設計されたバインダーが人間由来のサンプルにおいて機能的有用性を示したことで、アレルギー治療における応用の可能性を強く裏付けられました。
その他にも、既知ホモログを持たない新規タンパク質BBF-14や核酸結合ヌクレアーゼ、AAVリターゲティング等多様なターゲットに対し、高親和性バインダーを設計しました。BindCraftは、12種の多様なターゲットに対するバインダー設計能力を実証されています。設計されたバインダーの親和性はほとんどがナノモルの範囲でした。実験的な成功率は10~100%(平均46.3%)であり、RFdiffusionなどの既存のパイプラインと比較しても、実験的スクリーニングの必要性を大幅に削減する成果をあげています。
5. まとめ
BindCraftは、AF2の革新的な能力を最大限に引き出し、機能性タンパク質バインダーの計算設計における”one design, one binder”という目標に大きく近づく進歩を遂げています。計算専門知識が限られた研究者でも、オンデマンドで高親和性バインダーを設計できる道を示しています。
現時点では、AF2ネットワークを介した誤差逆伝播法は大量のGPUメモリを必要とし、大規模なタンパク質設計では課題が生じる可能性があります。また、AF2 monomerモデルによるフィルタリングが、真に高親和性なバインダーを除外したり、偽陽性を予測したりする可能性も指摘されています。これは、in silicoの指標が必ずしも実験的な結合親和性を正確に予測できないという、計算タンパク質設計における共通の課題でもあります。
BindCraftの成功率は非常に高く、開発にかかる時間やコストの大幅な削減へは大きく期待できるものと考えられます。また、かなり多様なターゲットタンパク質についての検証もされており、今まで標的とすることが難しかった対象への拡大が期待できると考えられます。特に、アレルゲンへのバインダー設計から、ヒト由来細胞での高い効果が確認されており、多種多様なアレルゲンへと対象を広げて、さらなる検証も期待したいです。アレルギー関連の患者は多く、この領域での早期の実用化は、多くの方々の安心した生活に貢献するものと思われます。


コメント