多くの製品があって、どの製品とどの製品が似ているのか、似ていないのか?などを調べるにはどうすればよいでしょう?
似ているもの同士を集めたり、似ていないものと区別するには、クラスター分析が大変役に立ちます。
例えば、ワインの成分情報から、クラスター分析を用いてワインの分類をしてみましょう!


1.クラスター分析とは
サンプルを似たもの同士を集団としてグループ化するのが、クラスター分析です。
与えられたサンプルをラベル情報なしに自動的に分類することができる手法で、教師なし学習になります。
階層クラスター分析とは、サンプル間の距離に基づいて、最も似ているものを集めてクラスターを作っていく方法です。
階層的なツリー構造(樹形図、デンドログラム)を構築し、グループ間の類似性を可視化することができます。
サンプル数が多いと、非階層クラスター分析が使われることが多いです。
https://hira-labo.com/archives/1969
今回は階層クラスター分析について記載していきます。
2.階層クラスター分析(データセットwine.dataを使用)
今回は、ワインの成分情報を用いて、ワインを分類してみます。
ワインの使用データセットはwine.data(UC Irvine Machine Learning Repository保有)です。
wine.dataには、178種のwineの情報が含まれていて、各ワインの情報としては、以下の13項目あります。
1) Alcohol(アルコール)、 2) Malic acid(リンゴ酸)、 3) Ash(灰分)、 4) Alcalinity of ash(灰のアルカリ度)、 5) Magnesium(マグネシウム)、 6) Total phenols(総フェノール量)、 7) Flavanoids(フラバノイド)、 8) Nonflavanoid phenols(非フラバノイドフェノール類)、 9) Proanthocyanins(プロアントシアニン)、 10)Color intensity(色の濃さ)、 11)Hue(色相)、 12)OD280/OD315 of diluted wines(希釈ワインのOD280/OD315)、 13)Proline(プロリン)
178種の中から、上から30のデータに絞り、階層クラスター分析を実施してみます。
library(stats) #1 パッケージ
wine_data <- read.csv("http://archive.ics.uci.edu/ml/machine-learning-databases/wine/wine.data", header = F) #2 wine_dataを読み込む
head(wine_data) #3 V2以降が上記13項目の列
# V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13 V14
#1 1 14.23 1.71 2.43 15.6 127 2.80 3.06 0.28 2.29 5.64 1.04 3.92 1065
#2 1 13.20 1.78 2.14 11.2 100 2.65 2.76 0.26 1.28 4.38 1.05 3.40 1050
#3 1 13.16 2.36 2.67 18.6 101 2.80 3.24 0.30 2.81 5.68 1.03 3.17 1185
#4 1 14.37 1.95 2.50 16.8 113 3.85 3.49 0.24 2.18 7.80 0.86 3.45 1480
#5 1 13.24 2.59 2.87 21.0 118 2.80 2.69 0.39 1.82 4.32 1.04 2.93 735
#6 1 14.20 1.76 2.45 15.2 112 3.27 3.39 0.34 1.97 6.75 1.05 2.85 1450
wine_data2<-wine_data[1:30, 2:14] #4 30サンプルにデータを絞る。V1は分類番号なので削除。
result <- hclust(dist(wine_data2), method = "average") #5 距離構造のデータ(デフォルト"euclidean" ユークリッド距離)、methodは方法を指定(average群集法を指定)。
#デンドログラム作成(縦)
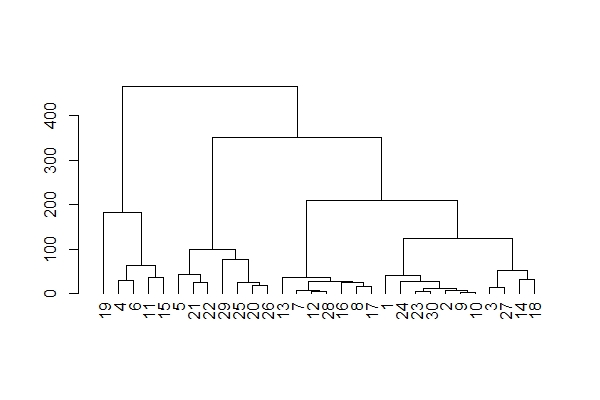
plot(as.dendrogram(result)) #6 デンドログラムのプロット①
rect.hclust(result, k = 3) #7 分類数をk=3と指定し、①に分類ごとのデフォルト赤(,border = ""で色指定可)で囲む。
#ラベルサイズの変更
par(cex=0.6) #ラベルサイズを指定した後 plot関数を実施
#5 dist(data, method=”@”) 個体同士の距離構造のデータで、methodをいれないとデフォルトの”euclidean”。@には”euclidean”(ユークリッド距離), “manhattan”(マンハッタン距離), “binary”(バイナリ距離), または “minkowski”(ミンコフスキー距離)を選択していれてください。
hclust(dist(wine_data2), method = “@”) クラスター間距離構造の手法を指定し、階層的クラスター分析を行います。 @には、”ward.D2″(ウォード法), “single”(最短距離法), “complete”(最長距離法), “average” (群平均法), “centroid” (重心法)を選択して入れてください。
#横向きデンドログラム
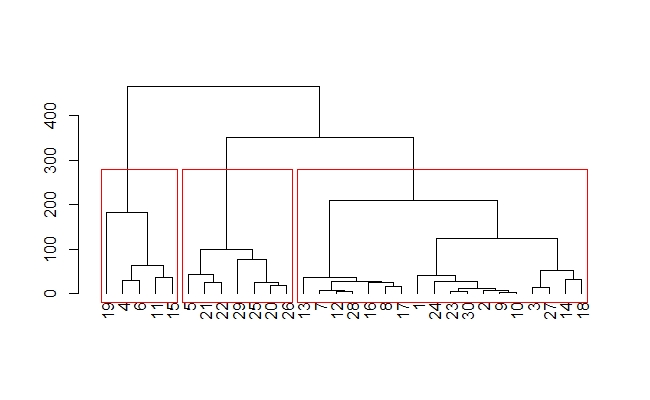
plot(as.dendrogram(result),horiz = TRUE) #8 横向きデンドログラムのプロット②
library(dendextend)
rect.dendrogram(as.dendrogram(result),k=3,horiz=TRUE) #9 分類数をk=3と指定し、②に分類ごとのデフォルト赤(,border = ""で色指定可)で囲む。
#8, 9で横向きのデンドログラムと、各クラスターを赤枠で囲むことができます。
#クラスターごとに枝の色変える
plot(set(as.dendrogram(result),'branches_k_color', k=3)) #10 デンドログラムの枝の色を変える。
plot(set(as.dendrogram(result),'branches_k_color', k=3), horiz=TRUE) #11 デンドログラムの枝の色を変えて、横向きにする。
rect.dendrogram(as.dendrogram(result),k=3,horiz=TRUE)
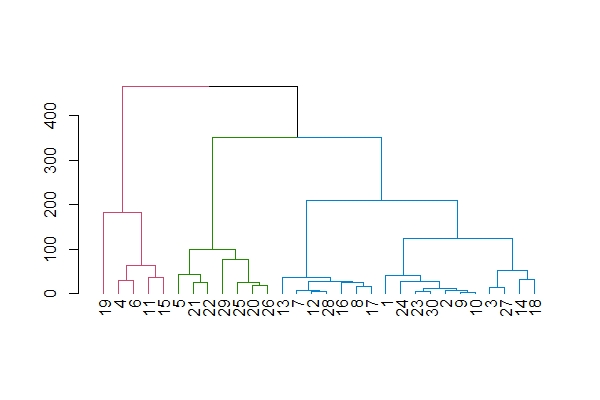
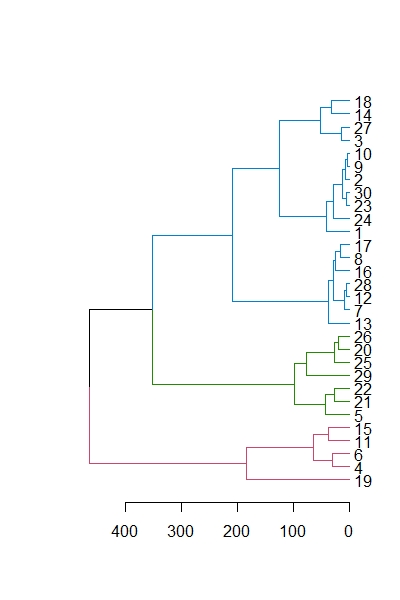
#10, 11 クラスターごとに枝の色を変えることができます。horiz=TRUEで横向きデンドログラムにできます。
3.シルエット分析(いくつに分類するのがよいか)
シルエット分析は、クラスタリング結果の指標として用いられる手法です。
この方法は、各サンプルがどの程度他のクラスタと密接に関連しているか、またどの程度独立しているかを評価することができます。
シルエット値は、各サンプルが所属するクラスタ内で他のサンプルとの距離と、他のクラスタとの距離を比較して求められます。
値の範囲は-1から1で、値が大きいほど、クラスタ内の各サンプルの関連性が高く、他のクラスタとの分離性が高いことを示します。
#シルエット分析
v_sil <- numeric(nrow(wine_data2)) #wine_data2の列(サンプル)数のカラムの作成
library(cluster)
for(i in 2:(nrow(wine_data2) - 1)){
sil <- silhouette(cutree(result, k = i), dist(wine_data2))
v_sil[i] <- summary(sil)$avg.width} #シルエット分析。クラスター内の距離情報と、クラスター間の距離情報から算出
v_sil
#[1] 0.00000000 0.49152034 0.56442493 0.64721718 0.64071311 0.68443923 0.66172176 0.65291975
#[9] 0.62346770 0.59865823 0.56841502 0.51544628 0.47545777 0.37283868 0.34517966 0.32728805
#[17] 0.34868349 0.32738313 0.27876591 0.25384855 0.25250787 0.23810427 0.21861302 0.17343921
#[25] 0.16321502 0.11275866 0.09261269 0.07132765 0.03668876 0.00000000
which.max(v_sil) #シルエット値の最大値の分類数
#[1] 6 # 分類数6が最適
#シルエット分析可視化
plot(1: nrow(wine_data2), v_sil, type="h") #シルエット分析の可視化
best <- which.max(v_sil)
axis(1, best, paste("最適数", best, sep = "\n"), col = "red", col.axis = "red") #シルエット分析結果に最適数を記入
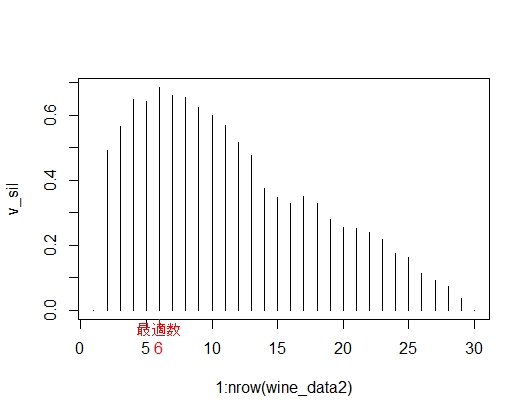
シルエット値が最大になるのは、分類数6のときであることが分かりました。
#最適分類数でのデンドログラム作成
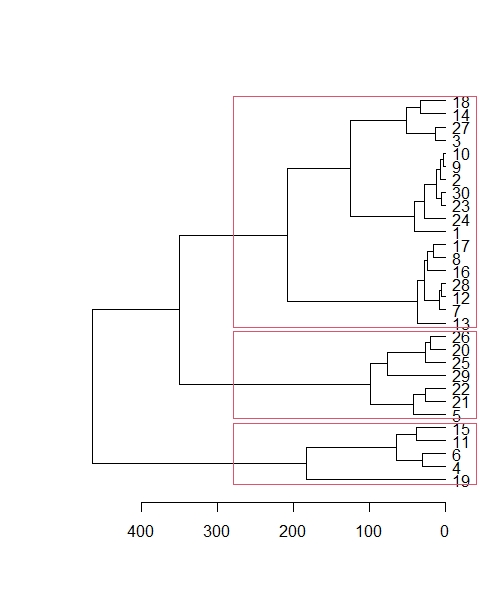
plot(set(as.dendrogram(result),'branches_k_color', k=6)) #k=に分類数
rect.hclust(result, k = 6, border = "gray")
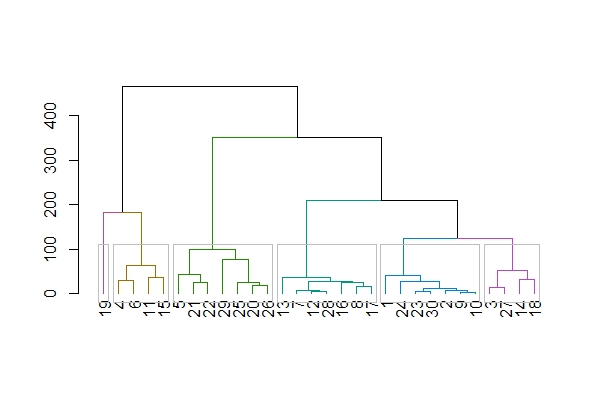
wine_dataの30サンプルは、与えられた成分情報から6つにクラスタリング(分類)することができました。
階層クラスター分析を通じて、ワインの成分データから似た者同士のグループ分けをおこないました。ぜひ、他のデータにも応用してみてください。
参考にしていただければ幸いです。
参考書籍▼▼





コメント