ノモグラムは、医療分野で患者の生存率予測や疾患リスク評価に広く活用されている視覚的な予測ツールです。
本記事では、コックス回帰を用いたノモグラムの作成手順を解説し、実際のデータを使って1年・2年後の生存率を予測する方法を紹介します。
1. ノモグラムとは
ノモグラムは、1997年の前立腺がんのリンパ節転移の予測に応用されて以降(※1)、がん患者の生存率予測や疾患の再発リスク評価に頻繁に活用され、臨床意思決定などでも重要な役割を果たします。
2000-2021年のノモグラムについてまとめたレビューでは、ノモグラムの研究報告は2018年以降急速に増加し、特に中国やアメリカから多く発表されているとのことです(※2)。主に腫瘍、特に泌尿器系の関連報告が多いようです。
ノモグラムの最大の特徴は、複数の予測因子(例:年齢、腫瘍ステージ、バイオマーカー)を直感的に捉えやすいスコアに変換し、総合的なリスク評価(例:1年後生存率、2年後生存率)が可能なところです。
これにより、数値的な予測モデルと異なり、医師や患者が複数の因子がどの程度予後に寄与するのかなどが視覚的に理解しやすく、よりよい治療について円滑な意思決定に繋げることができます。
また、各個人の複数因子によって臨床転帰を予測できるため、個別化された治療に貢献することができます。
➢ シンプルで一般の人にもわかりやすい(⇒ 医師と患者で円滑に情報のやりとりがしやすい)
➢ 疾患に関わる複数の因子によって、予後が予測できる(⇒ 患者の因子によって予測が可能)
主にコックス回帰やロジスティック回帰による予後予測を視覚化するのに使われています。
2. ノモグラムを書いてみよう(コックス回帰)
以前投稿した、コックス回帰におけるノモグラムを作成してみます。
前回同様、survival パッケージに入っているlung(進行肺がん患者の生存率)のデータセットを使用します。
inst: 機関コード、time: 生存期間(日)、status: 打ち切りステータス 1=打ち切り, 2=死亡、
age: 年齢(歳)、sex: 性別: 男性=1 女性=2、
ph.ecog: ECOGパフォーマンスステータス(身体機能レべルの指標 医師の診断)0=無症状、1=症状はあるが完全に歩行可能、2=1日の50%未満はベッドにいる、3=1日の50%以上はベッドにいるが寝たきりではない、4=寝たきり、
ph.karno: KSPスコア(身体機能レベルの指標 医師の診断)(悪い=0-良い=100)、
pat.karno: KSPスコア(身体機能レベルの指標 患者の診断)、meal.cal: 食事時の消費カロリー、
wt.loss: 過去6ヶ月間の体重減少(ポンド)
今回は、年齢、性別、ECOGパフォーマンスステータスから、1年後生存率、2年後生存率を求めるノモグラムを作成します。
##必要なパッケージ(インストールがまだの場合は、install.packages()を実行)
library(survival)
library(rms)
#lungデータの確認
head(lung)
# inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
#1 3 306 2 74 1 1 90 100 1175 NA
#2 3 455 2 68 1 0 90 90 1225 15
#3 3 1010 1 56 1 0 90 90 NA 15
#4 5 210 2 57 1 1 90 60 1150 11
#5 1 883 2 60 1 0 100 90 NA 0
#6 12 1022 1 74 1 1 50 80 513 0
#欠損値の削除
lung_n <- na.omit(lung)
head(lung_n)
# inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
#2 3 455 2 68 1 0 90 90 1225 15
#4 5 210 2 57 1 1 90 60 1150 11
#6 12 1022 1 74 1 1 50 80 513 0
37 7 310 2 68 2 2 70 60 384 10
#8 11 361 2 71 2 2 60 80 538 1
#9 1 218 2 53 1 1 70 80 825 16
#変数の分布情報を設定して以降の解析で使用
dd <- datadist(lung)
options(datadist = "dd")
#性別を数値から名称に変更して、より視覚的に捉えやすくする
lung_n$sex <- factor(lung_n$sex, levels = c(1, 2), labels = c("Male", "Female"))
#コックス回帰
cox_model <- cph(Surv(time, status) ~ age + sex + ph.ecog, data = lung_n, x = TRUE, y = TRUE, surv = TRUE)
surv_fit <- survfit(cox_model)
#コックス回帰の半年後、1年後、2年後の生存率予測を確認する。
summary(surv_fit, times = c(180, 365, 730))
#Call: survfit(formula = cox_model)
#
# time n.risk n.event survival std.err lower 95% CI upper 95% CI
# 180 121 43 0.753 0.0335 0.6899 0.821
# 365 49 44 0.417 0.0431 0.3405 0.511
# 730 10 29 0.116 0.0329 0.0669 0.203
#ノモグラム作成
nom <- nomogram(cox_model,
fun = list(Surv1year=function(x) surv(1 * 365, x), # 1年生存率
Surv2year=function(x) surv(2 * 365, x)), # 2年生存率
lp = FALSE) #生存率だけ表示
plot(nom)
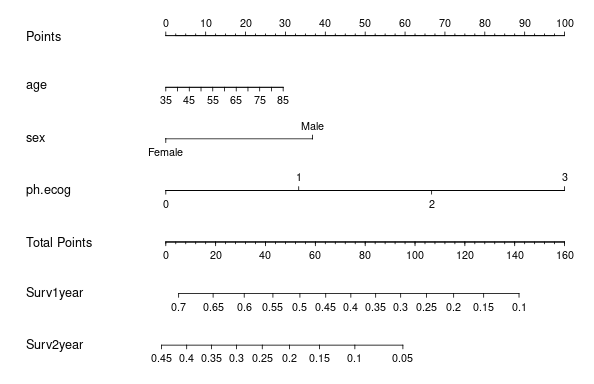
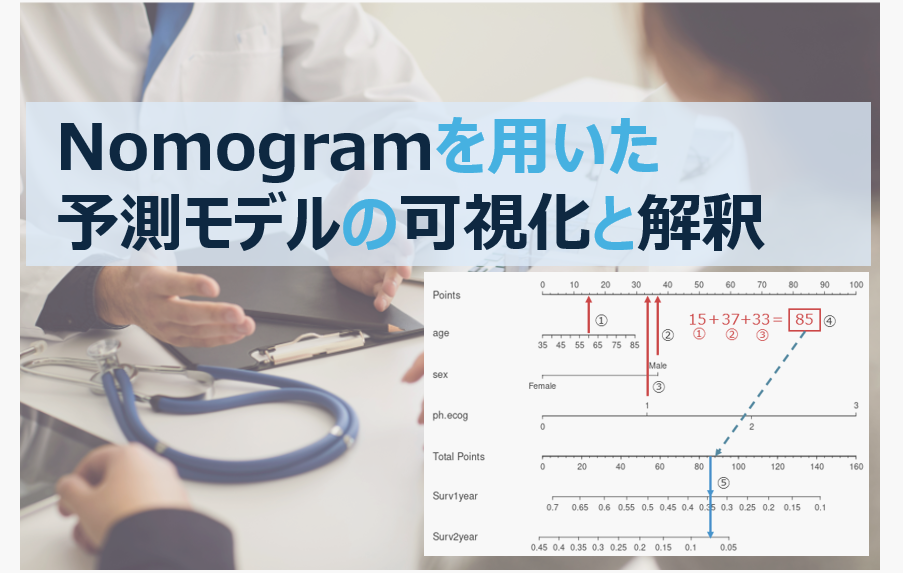
ノモグラムが作成できました。
3. ノモグラムの解釈
ノモグラムのリスクスコアの読み方について解説します。
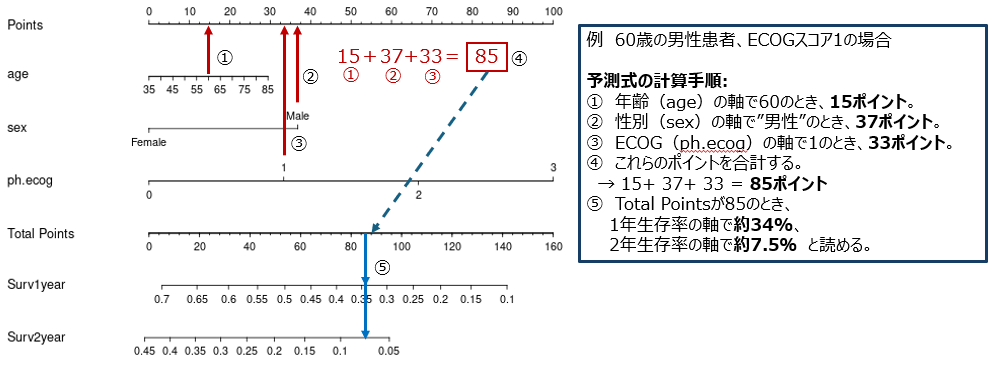
60歳男性患者で、ECOGスコア1の場合、1年生存率は約34%、2年生存率は約7.5%であることが分かりました。
このように、現在の患者の状況をスコア化して、予測を確認することが可能です。
ノモグラムは、複数の予測因子を統合し、直感的に予後を評価できる強力なツールです。本記事では、コックス回帰を活用してノモグラムを作成し、その解釈方法について説明しました。
参考論文
※1 Puppo P, Perachino M. Clinical stage, prostate-specific antigen and Gleason grade to predict extracapsular disease or nodal metastasis in men with newly diagnosed, previously untreated prostate cancer. A multicenter study. A. Ur. O. Cooperative Group. Eur Urol. (1997) 32:273–9. doi: 10.1159/000480824
※2 Wang X, Lu J, Song Z, Zhou Y, Liu T and Zhang D (2022) From past to future: Bibliometric analysis of global research productivity on nomogram (2000–2021). Front. Public Health 10:997713. doi: 10.3389/fpubh.2022.997713

生存時間解析について、今までカプランマイヤー曲線、コックス比例ハザードモデルなどについてRのコードも含めた解説を行いましたので、参考にしていただけると幸いです。



コメント