前回(情報Ⅰプログラミング×化学①)、Pythonで化学構造の表現形式SMILESと化学構造を扱うライブラリRDKit、そして化学データベースPubChemを使って、パソコンの上に分子の構造式を描画する方法を紹介しました。

次は、Pythonで分子がどんな性質を調べていきます💡
Pythonで、分子の分子量や水と油への馴染みやすさ(LogP)、隣り合う分子同士が引き合いやすいか(水素結合の数)などの性質を調ることができます。
これらの性質は、新しい「飲み薬」の候補を効率よく見つけ出すときに、とても役立つんです💡
リピンスキーの法則といって、体に吸収されやすく、効果を発揮しやすい薬の候補となる分子が、これらの性質においてどのような特徴を持っているかの目安を示しています。
プログラミングでその世界を少し覗いて、未来の薬づくりを体験してみましょう!
1. 分子の性質(分子量、水素結合ドナー・アクセプター数、LogP)
2. Pythonで分子の性質を計算する準備
3. 分子量の計算
4. 水素結合ドナー・アクセプター数の計算
5. LogPの計算
6. 他の計算で求めたLogP、実測値との比較
7. まとめ
1. 分子の性質(分子量、水素結合ドナー・アクセプター数、LogP)
今回はRDKitやPubChemPyのライブラリを使って、分子の性質を調べていきます。
今回は新しい医薬品を作る際に、その候補成分として着目されることがある、以下の4つの性質をPythonで情報を集めたいと思います💡
・分子量: 分子に含まれる全ての原子の原子量の総和です。この原子量とは、12C(質量数12の炭素原子)の質量を12と定め、これを基準として表した相対的な質量のことです。
・水素結合ドナー数: 分子の中で、他の分子に水素原子を提供して水素結合を作ることができる部分の数です。主に-OH基や-NH基などが該当します。
・水素結合アクセプター数: 分子の中で、他の分子から水素原子を受け取って水素結合を作ることができる部分の数です。主に酸素原子(O)や窒素原子(N)が該当します。
 ・LogP (分配係数): オクタノール/水 分配係数の常用対数値のことです。これは分子が水と油のどちらに馴染みやすいかの度合いを示す数値指標で、LogPの値が大きいほど油に溶けやすく、小さいほど水に溶けやすい傾向があります。
・LogP (分配係数): オクタノール/水 分配係数の常用対数値のことです。これは分子が水と油のどちらに馴染みやすいかの度合いを示す数値指標で、LogPの値が大きいほど油に溶けやすく、小さいほど水に溶けやすい傾向があります。
💡分子量(500以下)、水素結合ドナー(5個以下)・アクセプター数(10個以下)、LogP(5以下)は、経口医薬品になりやすい化合物の特徴として(Lipinski(リピンスキー)の法則)知られています。
この法則にも諸説ありますが、この条件に当てはまる化合物を探してみるのも面白いですね^^
2. Pythonで分子の性質を計算する準備
Pythonを使って分子の性質を計算する準備を始めます。
Google Colaboratory (Colab) などのPython実行環境を使います。
◆Step 1:必要なライブラリをインストール&インポート
今回使うRDKitとPubChemPy(PubChemから情報を取得するためのライブラリ)をインストールします。Colabでは、以下のコードをセルに入力して実行してください。(もし既にインストール済みであれば、このステップは不要です。)
!pip install rdkit-pypi pubchempy
Pythonでこれらのライブラリを使えるようにします。今回は、性質計算に使うRDKitのモジュールも一緒にインポートします。
from rdkit import Chem from rdkit.Chem import Draw # 分子構造の描画用 from rdkit.Chem import Descriptors # 分子量やLogPなどの計算用 from rdkit.Chem import Lipinski # 水素結合ドナー/アクセプター数などを計算用 import pubchempy as pcp #Jupyter NotebookやGoogle Colabで構造をきれいに表示 from rdkit.Chem.Draw import IPythonConsole IPythonConsole.ipython_useSVG=True # SVG形式で表示(推奨)
◆Step 2:調べたい化合物名を入力しよう!
次に、性質を調べたい化合物の名前(英語名)を指定します。
今回は、カフェイン(コーヒーにも含まれ、眠気防止や痛み止めにも使用される成分)、アスピリン(痛み止めとして使用される成分)、オレイン酸(食用油として使用される)の3つについて調べていきます。
# ここに調べたい化合物の英語名を入力してください compound_name = "caffeine"
上記のコードの “caffeine” の部分を、調べたい化合物の英語名に書き換えると、以降のコードで、調べたい成分の情報を得ることができます。
“aspirin”(アスピリン)、”oleic acid”(オレイン酸)、”ethanol”(エタノール)、”water”(水)など
◆Step 3:PubChemから分子情報を取得し、RDKitで扱える形に!
指定した化合物名を使って、PubChemデータベースからその分子のSMILESを取得します。そのSMILESを元にRDKitが扱える分子オブジェクトを作成します。この分子オブジェクトで、様々な性質を計算していきます。
mol = None # RDKitの分子オブジェクトを格納する変数 (最初は空)
smiles = None # SMILES文字列を格納する変数 (最初は空)
try:
# PubChemで化合物名を検索
compound_data_list = pcp.get_compounds(compound_name, 'name')
if compound_data_list: # もし化合物が見つかったら
pubchem_compound = compound_data_list[0] # 最初の検索結果を使う
# SMILES文字列を取得 (isomeric_smilesがあればそれを、なければcanonical_smiles)
smiles = pubchem_compound.isomeric_smiles if pubchem_compound.isomeric_smiles else pubchem_compound.canonical_smiles
if smiles: # SMILESが取得できたら
print(f"取得したSMILES: {smiles}")
# SMILESからRDKitの分子オブジェクトを作成
mol = Chem.MolFromSmiles(smiles)
if mol: # 分子オブジェクトがちゃんとできたら
print(f"'{compound_name}' の分子オブジェクト作成に成功しました!")
# 分子構造も表示してみましょう
display(Draw.MolToImage(mol, size=(300,300))) # Colab/Jupyter環境で表示
else:
print(f"エラー: SMILES '{smiles}' から分子オブジェクトを作成できませんでした。")
else:
print(f"エラー: '{compound_name}' のSMILES情報がPubChemで見つかりませんでした。")
else:
# PubChemで指定された化合物名が見つからなかった場合
print(f"エラー: '{compound_name}' がPubChemデータベースで見つかりませんでした。英語の名称か、スペルを確認してください。")
except Exception as e:
print(f"処理中に予期せぬエラーが発生しました: {e}")
# 出力結果 取得したSMILES: CN1C=NC2=C1C(=O)N(C(=O)N2C)C 'caffeine' の分子オブジェクト作成に成功しました!
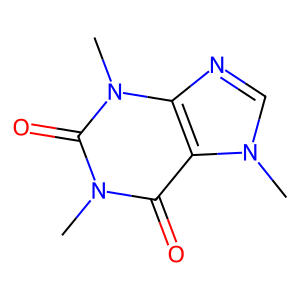
このコードを実行すると、指定した化合物のSMILESと、その構造式(画像)が表示されます。
もしエラーが出た場合は、エラーメッセージをよく読んで、化合物名が正しいか、インターネットに繋がっているかなどを確認してみてください。
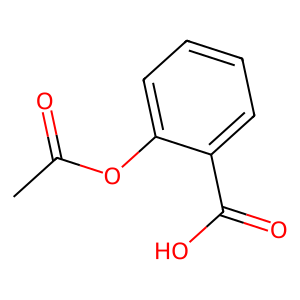
・compound_name =aspirinの場合
取得したSMILES: CC(=O)OC1=CC=CC=C1C(=O)O
‘aspirin’ の分子オブジェクト作成に成功しました!
 ・oleic acidの場合
・oleic acidの場合
取得したSMILES: CCCCCCCC/C=C\CCCCCCCC(=O)O
‘oleic acid’ の分子オブジェクト作成に成功しました!
3. 分子量の計算
作成した分子オブジェクト (mol) を使って、RDKitのDescriptors.MolWt() で分子量を計算します。
# caffeineの場合 Descriptors.MolWt(mol)
# 出力結果 194.194
・aspirinの場合: 180.15899999999996
・oleic acidの場合: 282.4679999999999
4. 水素結合ドナー・アクセプター数の計算
水素結合は、分子同士が引き合う比較的作用の弱い結合の一つですが、水の性質やタンパク質の形など、化学や生物の世界で非常に重要な役割を果たしています。
RDKitを使うと、分子がどれくらい水素結合を作りやすいかの目安となる数を計算できます。
水素結合ドナー (Hydrogen Bond Donor): 分子の中で、水素結合のために水素原子をあげる供与することができる部分の数。OH, NHの数。
水素結合アクセプター (Hydrogen Bond Acceptor): 分子の中で、水素結合のために水素原子を供与されることができる部分の数。O, N原子の数。
これらは、Lipinski モジュールの中の関数で計算できます。
num_h_donors = Lipinski.NumHDonors(mol)
num_h_acceptors = Lipinski.NumHAcceptors(mol)
print(f"水素結合ドナーの数 ({compound_name}): {num_h_donors}")
print(f"水素結合アクセプターの数 ({compound_name}): {num_h_acceptors}")
# 出力結果 水素結合ドナーの数 (caffeine): 0 水素結合アクセプターの数 (caffeine): 6
・aspirinの場合:
水素結合ドナーの数 (aspirin): 1
水素結合アクセプターの数 (aspirin): 3
・oleic acidの場合:
水素結合ドナーの数 (oleic acid): 1
水素結合アクセプターの数 (oleic acid): 1
5. LogPの計算(SLogP)
分配係数LogPは、分子が水と油(1-オクタノールという油で代用)のどちらに溶けやすいかの指標です。
LogPが大きいほど油に溶けやすく(脂溶性が高い)、小さいほど水に溶けやすい(水溶性が高い)傾向があります。薬が体の中で吸収されやすかったり、細胞膜を通り抜けやすかったりする性質にも関わるため、医薬品開発でとても重要視される値の一つです。
RDKitでは、Descriptors.MolLogP() で計算できます。
なお、RDKitでは、Crippen法でのLogPの計算をしています(SLogP)。原子あるいは小さな原子グループごとの貢献度を足し合わせるような計算方法です。
log_p_value = Descriptors.MolLogP(mol)
print(f"計算されたLogP ({compound_name}): {log_p_value:.2f}")
# 出力結果 計算されたLogP (caffeine): -1.03
・aspirinの場合:
計算されたLogP (aspirin): 1.31
・oleic acidの場合:
計算されたLogP (oleic acid): 6.11
6. 他の計算で求めたLogP(XLogP)、実測値との比較
◆XLogP
PubChemのデータベースには、RDKitのCrippen法とは異なるXLogP(XLogP3)という計算方法でLogPを求めた計算値が掲載されています。これは、原子タイプごとの貢献度に、様々な補正(分子内の原子同士の影響など)を加えて予測精度を上げる、比較的新しい手法です。
pubchem_xlogp = "N/A"
try:
# 化合物名でPubChemを検索し、Compoundオブジェクトを取得
compounds = pcp.get_compounds(compound_name, 'name')
if compounds:
target_compound_obj = compounds[0]
# Compoundオブジェクトの xlogp 属性を取得
if hasattr(target_compound_obj, 'xlogp') and target_compound_obj.xlogp is not None:
pubchem_xlogp = f"{float(target_compound_obj.xlogp):.2f}" # 小数点以下2桁で表示
else:
pubchem_xlogp = "xlogp属性なし"
print(f"PubChemの計算値 (XLogP3相当): {pubchem_xlogp}")
else:
print(f"PubChemで {compound_name} が見つからず、比較できませんでした。")
except Exception as e:
print(f"PubChemからの情報取得中にエラーが発生しました: {e}")
# 出力結果 PubChemの計算値 (XLogP3相当): -0.10
aspirinの場合:
PubChemの計算値 (XLogP3相当): 1.20
oleic acidの場合:
PubChemの計算値 (XLogP3相当): 6.50
これらの計算から、SLogとXLogPなど計算方法によって、数値が異なることが分かりました。
◆計算値と実測値の比較
では、実際の実測値と比較してみます!実測値は、既報などからPubChemのサイトにも記載されています。
参考)https://pubchem.ncbi.nlm.nih.gov/compound/Caffeine
https://pubchem.ncbi.nlm.nih.gov/compound/Oleic-Acid
https://pubchem.ncbi.nlm.nih.gov/compound/Aspirin
| 化合物名 | RDKit (SLogP) 計算値 | PubChem (XLogP※1) 計算値 | 実測値 (PubChem掲載※2) |
|---|---|---|---|
| カフェイン (Caffeine) | -1.03 | -0.10 | -0.07 |
| アスピリン (Aspirin) | 1.31 | 1.20 | 1.18, 1.19 |
| オレイン酸 (oleic acid) | 6.11 | 6.50 | 7.64 |
Compoundオブジェクトのxlogp属性から取得した値です。これは多くの場合、XLogP3という計算手法による値に相当します。※2 PubChemのウェブサイトの「Experimental Properties」セクションに掲載されている代表的な実験値です。実験値は測定条件や文献により複数の報告がある場合があります。
以上より、計算値のSLogPもXLogPも実測値とは完全に一致はしないことが分かりますが、化合物の性質の傾向をつかむには計算値を参考にするのもよいかもしれません。
💡 なお。上述のリピンスキーの法則でいくと、オレイン酸はLogP値が5より高く、候補としては該当しない、と言えます。法則については、様々な考え方があります。
7. まとめ
今回はPythonとRDKit、PubChemを使い、分子が持つ様々な「性質」をコンピュータで計算・予測する方法を探求しました!
・化合物名からスタート: 化合物名さえ分かれば、PubChem経由で分子情報を取得し、RDKitで扱える形にできました。
・分子の性質を計算: RDKitを使って、分子の基本的な重さである分子量、他の分子とくっつきやすさの目安となる水素結合の数、そして水と油への馴染みやすさを示すLogPを計算・予測しました。
・計算値の比較と実測値: LogPでは、RDKit (SLogP) とPubChem (XLogP3相当) の計算値を比べ、さらにPubChemウェブサイトの実験値とも見比べることで、計算による予測の面白さと、方法による違いを実感できたと思います。
・医薬品開発へのつながり: 計算したこれらの性質が、新しいお薬の候補を見つける際のヒント(リピンスキーの法則など)にも関連していることを学びました。
プログラミングと化学が手を組むことで、コンピュータ上で分子の世界を探求できます。もちろん実験は重要ですが、多くの候補物質を一つ一つ実験で確かめるのは大変です。そこで今回体験したように、コンピュータで分子の性質を予測計算し、有望そうなものを見極めることで、研究はずっと効率的になります。
情報Ⅰで学ぶプログラミングの知識を活かして、自分だけの「化学探究」を進めてみてください!




コメント